O DNA inteiro de uma célula é replicado durante a fase S (síntese) do ciclo celular. O princípio da replicação é baseado no emparelhamento de pares de bases de nucleótidos complementares: a adenina forma pontes de hidrogénio com a timina (ou uracilo no RNA) e a guanina forma pontes de hidrogénio com a citosina. A replicação ocorre antes da divisão celular, na fase S do ciclo celular. Este processo permite a formação de 2 conjuntos de cromossomas durante a metáfase da mitose, sendo depois igualmente divididos em 2 novas células durante a separação (anáfase).
Última atualização: Dec 13, 2024
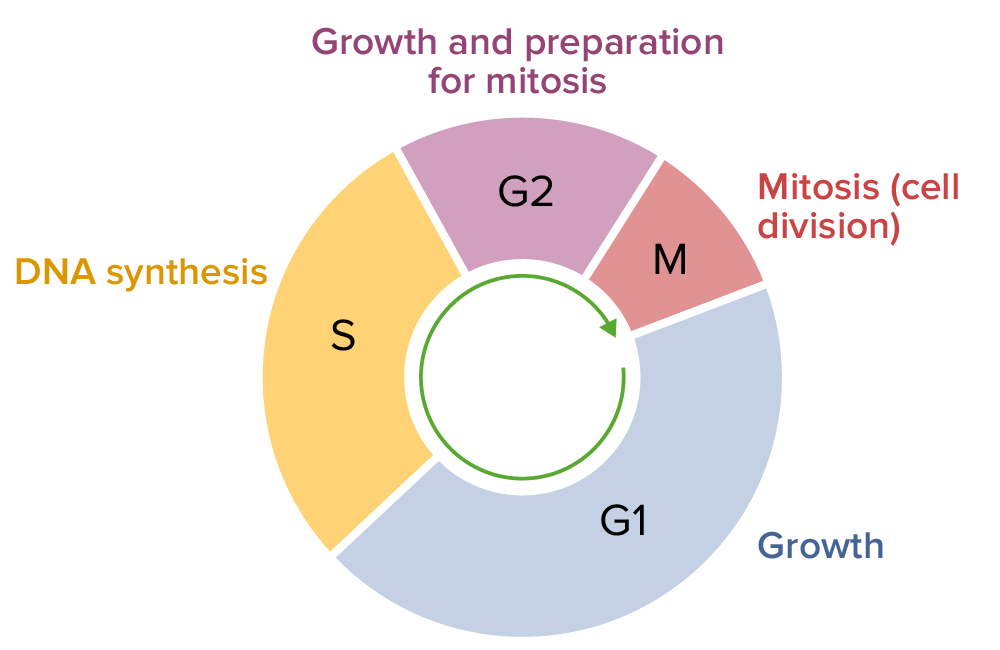
Ciclo celular, constituído pelas fases G1, S, G2 e M
Imagem pro Lecturio.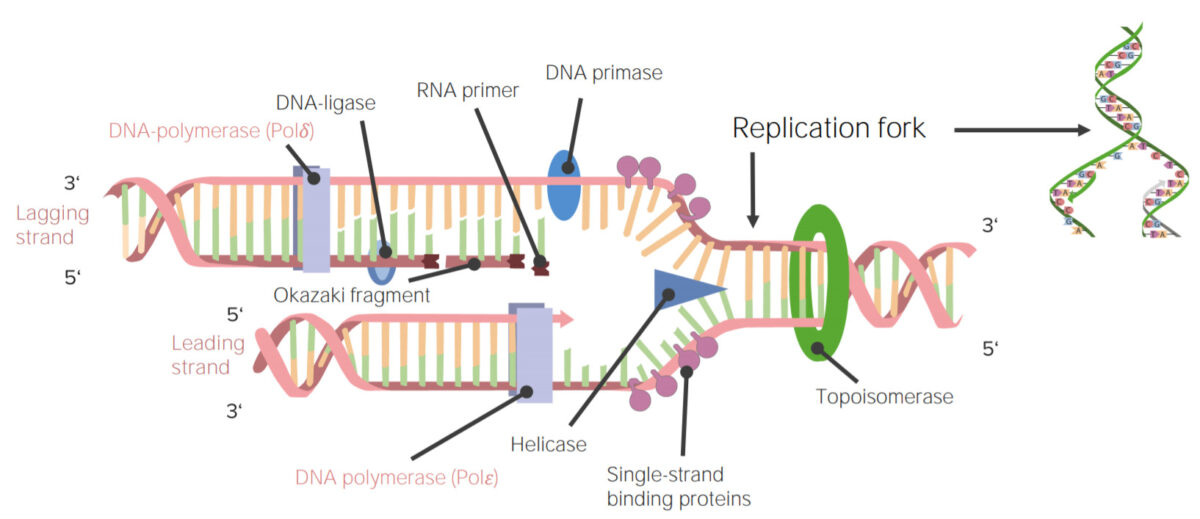
A replicação ou síntese de DNA é o processo da cópia de uma molécula de DNA de cadeia dupla. Este processo é fundamental para toda a vida tal como a conhecemos.
Imagem: “DNA Replication” by Mariana Ruiz. Licença: Public Domain| Polimerase | Função | Atividade da exonuclease |
|---|---|---|
| α | Sintetiza o primer de RNA e inicia a síntese do DNA ao longo da cadeia atrasada | 3′ → 5′ |
| β | Repara o DNA | Nenhuma |
| γ | Replica o DNA mitocondrial | 3′ → 5′ |
| δ | Sintetiza a cadeia atrasada, preenchendo as lacunas de DNA após a remoção do primer. | 3′ → 5′ |
| ε | Sintetiza a cadeia líder | 3′ → 5′ e 5′ → 3′ |