As modificações pós-transcripcionais (PTMs, pela sigla em inglês) são processos que facilitam a produção de RNA RNA A polynucleotide consisting essentially of chains with a repeating backbone of phosphate and ribose units to which nitrogenous bases are attached. RNA is unique among biological macromolecules in that it can encode genetic information, serve as an abundant structural component of cells, and also possesses catalytic activity. RNA Types and Structure maturado e funcional. Estes mecanismos regulatórios de resposta rápida permitem que diferentes proteínas sejam produzidas a partir de um gene Gene A category of nucleic acid sequences that function as units of heredity and which code for the basic instructions for the development, reproduction, and maintenance of organisms. Basic Terms of Genetics e atuam como reguladores do fenótipo e da taxa de proliferação. Estas modificações também podem desempenhar um papel em algumas formas de cancro e em doenças neurodegenerativas. O RNA RNA A polynucleotide consisting essentially of chains with a repeating backbone of phosphate and ribose units to which nitrogenous bases are attached. RNA is unique among biological macromolecules in that it can encode genetic information, serve as an abundant structural component of cells, and also possesses catalytic activity. RNA Types and Structure pré-mensageiro ( mRNA mRNA RNA sequences that serve as templates for protein synthesis. Bacterial mRNAs are generally primary transcripts in that they do not require post-transcriptional processing. Eukaryotic mRNA is synthesized in the nucleus and must be exported to the cytoplasm for translation. Most eukaryotic mRNAs have a sequence of polyadenylic acid at the 3' end, referred to as the poly(a) tail. The function of this tail is not known for certain, but it may play a role in the export of mature mRNA from the nucleus as well as in helping stabilize some mRNA molecules by retarding their degradation in the cytoplasm. RNA Types and Structure), chamado RNA RNA A polynucleotide consisting essentially of chains with a repeating backbone of phosphate and ribose units to which nitrogenous bases are attached. RNA is unique among biological macromolecules in that it can encode genetic information, serve as an abundant structural component of cells, and also possesses catalytic activity. RNA Types and Structure nuclear heterogéneo (hnRNA), é modificado pela adição de um cap 7-metillguanosina na extremidade 5' e uma cauda poli-A (poliadenilato) na extremidade 3' para estabilidade e proteção. Além disso, o hnRNA que contém intrões (sequências não codificantes) entre as sequências expressas, ou exões, sofre reparações (splicing). Este processo remove intrões para produzir um mRNA mRNA RNA sequences that serve as templates for protein synthesis. Bacterial mRNAs are generally primary transcripts in that they do not require post-transcriptional processing. Eukaryotic mRNA is synthesized in the nucleus and must be exported to the cytoplasm for translation. Most eukaryotic mRNAs have a sequence of polyadenylic acid at the 3' end, referred to as the poly(a) tail. The function of this tail is not known for certain, but it may play a role in the export of mature mRNA from the nucleus as well as in helping stabilize some mRNA molecules by retarding their degradation in the cytoplasm. RNA Types and Structure maturado com a sequência de codificação para tradução. O splicing alternativo, por outro lado, também exclui os intrões, mas formam-se várias combinações de exões, produzindo proteínas diferentes do mRNA mRNA RNA sequences that serve as templates for protein synthesis. Bacterial mRNAs are generally primary transcripts in that they do not require post-transcriptional processing. Eukaryotic mRNA is synthesized in the nucleus and must be exported to the cytoplasm for translation. Most eukaryotic mRNAs have a sequence of polyadenylic acid at the 3' end, referred to as the poly(a) tail. The function of this tail is not known for certain, but it may play a role in the export of mature mRNA from the nucleus as well as in helping stabilize some mRNA molecules by retarding their degradation in the cytoplasm. RNA Types and Structure original. Na edição de RNA RNA A polynucleotide consisting essentially of chains with a repeating backbone of phosphate and ribose units to which nitrogenous bases are attached. RNA is unique among biological macromolecules in that it can encode genetic information, serve as an abundant structural component of cells, and also possesses catalytic activity. RNA Types and Structure, a sequência de mRNA mRNA RNA sequences that serve as templates for protein synthesis. Bacterial mRNAs are generally primary transcripts in that they do not require post-transcriptional processing. Eukaryotic mRNA is synthesized in the nucleus and must be exported to the cytoplasm for translation. Most eukaryotic mRNAs have a sequence of polyadenylic acid at the 3' end, referred to as the poly(a) tail. The function of this tail is not known for certain, but it may play a role in the export of mature mRNA from the nucleus as well as in helping stabilize some mRNA molecules by retarding their degradation in the cytoplasm. RNA Types and Structure é alterada e difere do modelo de DNA DNA A deoxyribonucleotide polymer that is the primary genetic material of all cells. Eukaryotic and prokaryotic organisms normally contain DNA in a double-stranded state, yet several important biological processes transiently involve single-stranded regions. DNA, which consists of a polysugar-phosphate backbone possessing projections of purines (adenine and guanine) and pyrimidines (thymine and cytosine), forms a double helix that is held together by hydrogen bonds between these purines and pyrimidines (adenine to thymine and guanine to cytosine). DNA Types and Structure transcrito. O RNA RNA A polynucleotide consisting essentially of chains with a repeating backbone of phosphate and ribose units to which nitrogenous bases are attached. RNA is unique among biological macromolecules in that it can encode genetic information, serve as an abundant structural component of cells, and also possesses catalytic activity. RNA Types and Structure transportador e o RNA RNA A polynucleotide consisting essentially of chains with a repeating backbone of phosphate and ribose units to which nitrogenous bases are attached. RNA is unique among biological macromolecules in that it can encode genetic information, serve as an abundant structural component of cells, and also possesses catalytic activity. RNA Types and Structure ribossomal começam a partir de moléculas precursoras mais MAIS Androgen Insensitivity Syndrome longas e passam por etapas que incluem metilação, cortes e adição de nucleótidos.
Last updated: Apr 25, 2025
A informação genética do DNA DNA A deoxyribonucleotide polymer that is the primary genetic material of all cells. Eukaryotic and prokaryotic organisms normally contain DNA in a double-stranded state, yet several important biological processes transiently involve single-stranded regions. DNA, which consists of a polysugar-phosphate backbone possessing projections of purines (adenine and guanine) and pyrimidines (thymine and cytosine), forms a double helix that is held together by hydrogen bonds between these purines and pyrimidines (adenine to thymine and guanine to cytosine). DNA Types and Structure é copiada para o RNA RNA A polynucleotide consisting essentially of chains with a repeating backbone of phosphate and ribose units to which nitrogenous bases are attached. RNA is unique among biological macromolecules in that it can encode genetic information, serve as an abundant structural component of cells, and also possesses catalytic activity. RNA Types and Structure mensageiro ( mRNA mRNA RNA sequences that serve as templates for protein synthesis. Bacterial mRNAs are generally primary transcripts in that they do not require post-transcriptional processing. Eukaryotic mRNA is synthesized in the nucleus and must be exported to the cytoplasm for translation. Most eukaryotic mRNAs have a sequence of polyadenylic acid at the 3′ end, referred to as the poly(a) tail. The function of this tail is not known for certain, but it may play a role in the export of mature mRNA from the nucleus as well as in helping stabilize some mRNA molecules by retarding their degradation in the cytoplasm. RNA Types and Structure).
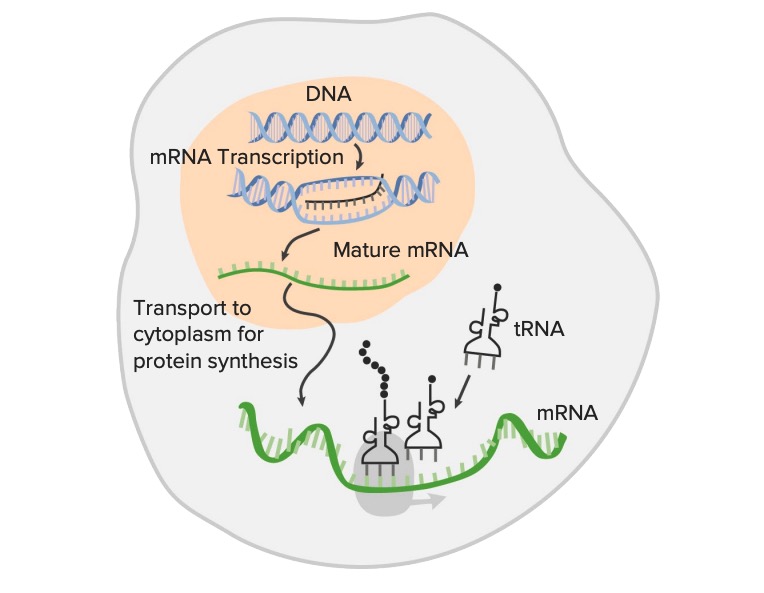
A expressão génica do DNA, a sequência genética, é transcrita para RNA (transcrição):
A transcrição da informação genética é o primeiro passo na expressão génica e é o processo pelo qual uma região codificante do DNA (estrutura de dupla cadeia) é usada como modelo para a síntese de RNA mensageiro (mRNA). O mRNA maturado é traduzido em aminoácidos, formando proteínas (tradução) com a ajuda do RNA ribossomal e RNA de transferência (tRNA). Esta imagem mostra a transcrição sem modificações pós-transcritas do RNA.
As transcrições primárias, ou produtos imediatos da transcrição, sofrem alterações para se tornarem biologicamente funcionais.
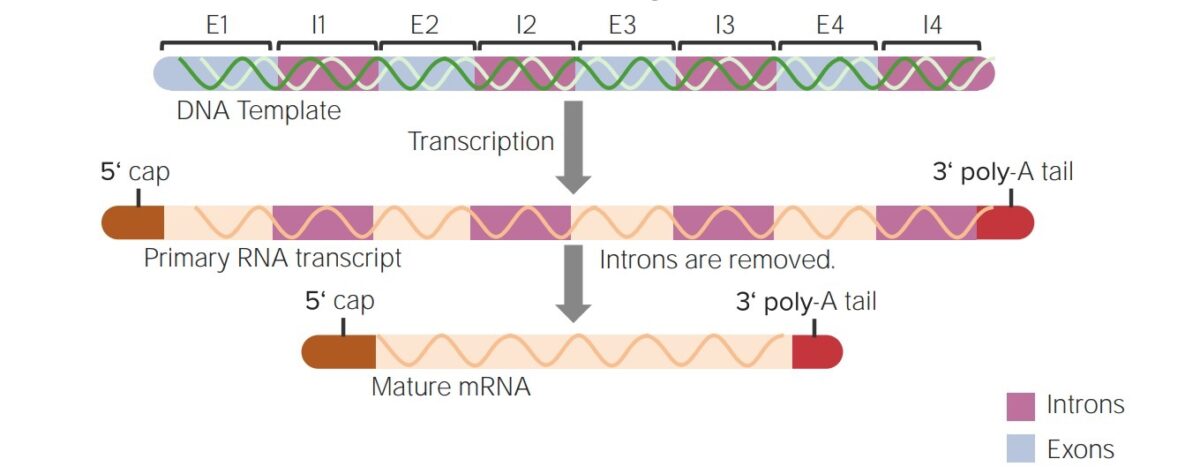
Resumo das modificações pós-transcricionais desde um hnRNA até um mRNA maduro:
A adição do cap 5′ e da cauda 3′ de poli-A e splicing (remoção das sequências intervenientes ou intrões)
7-Metilguanosina (resíduo de guanilil metilado) é adicionado à extremidade 5′ do hnRNA via:
Funções:
50 a 250 resíduos de adenilil (AMP pela sigla em inglês) são adicionados à extremidade 3′ do hnRNA via:
Função:
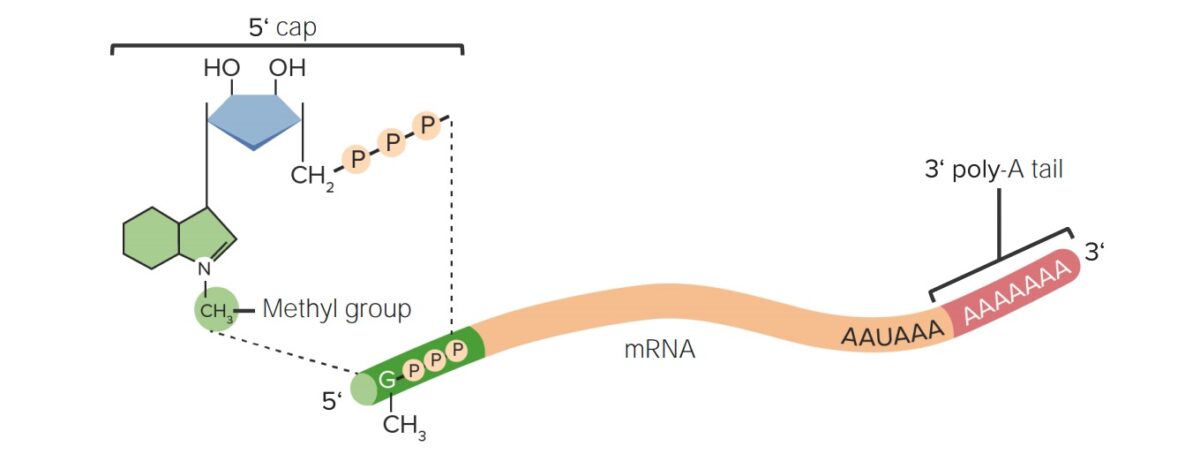
Modificações pós-transcricionais do RNA:
As modificações do cap 5′ (7-metilguanosina) e da cauda 3′ de poli-A evitam a degradação do mRNA no citosol.
RNA RNA A polynucleotide consisting essentially of chains with a repeating backbone of phosphate and ribose units to which nitrogenous bases are attached. RNA is unique among biological macromolecules in that it can encode genetic information, serve as an abundant structural component of cells, and also possesses catalytic activity. RNA Types and Structure heterogéneo nuclear (pré-mRNA) contém:
Processamento:
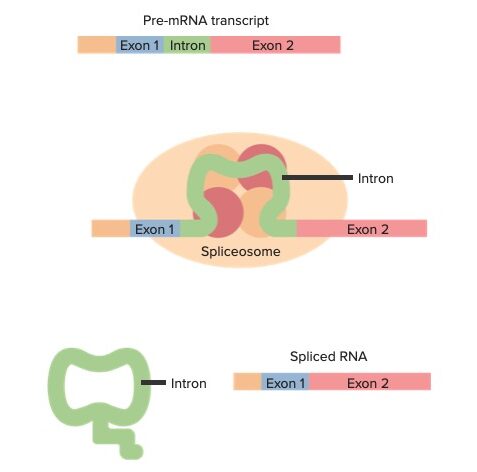
Exões e intrões pré-mRNA com um resumo do splicing (de cima para baixo):
A transcrição pré-mRNA contém exões e intrões. O transcrito interage com pequenas ribonucleoproteínas nucleares e outras proteínas, formando um spliceossoma em certas junções do transcrito. São feitos cortes nos locais de splicing e o intrão é libertado. Após sofrer splicing, o RNA passa a ter apenas exões, que contêm a sequência de codificação.
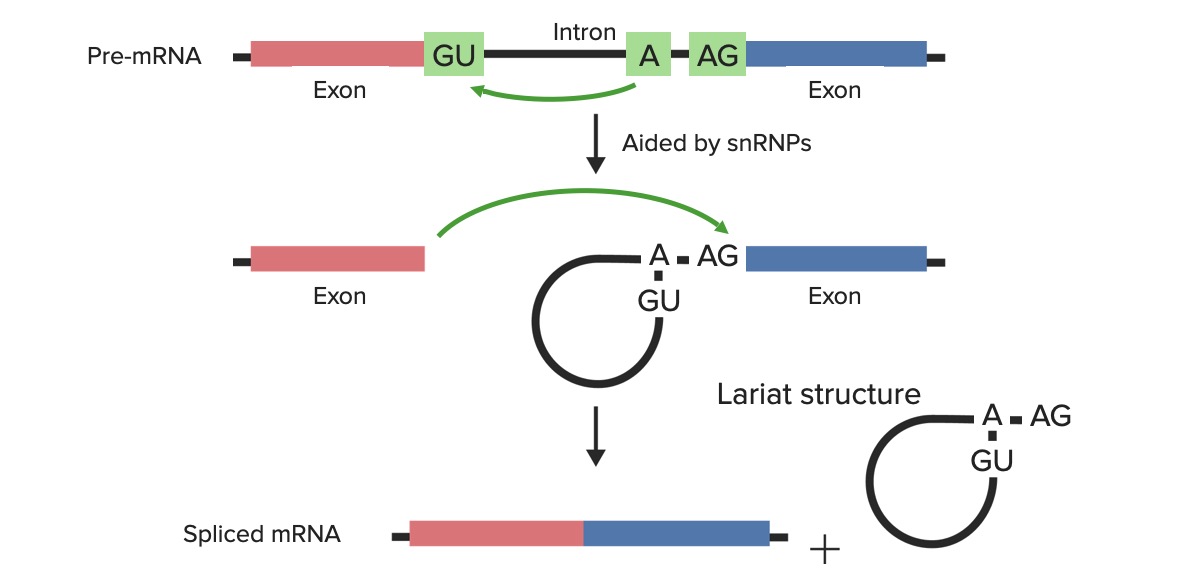
Aspetos técnicos do splicing:
Os pré-mRNA/hnRNA são compostos por exões e intrões. Pequenas ribonucleoproteínas nucleares + outras proteínas reconhecem o local da ramificação e as junções exão–intrão onde fazer o corte: o sítio dador 5′ (contendo a sequência GU invariável) e o sítio aceitador 3′ (contendo a sequência AG invariável). O transcrito de hnRNA + pequenas ribonucleoproteínas nucleares + outras proteínas combinam-se nestes locais e formam o spliceossoma.
Imagem de cima: Através da ajuda de pequenas ribonucleoproteínas nucleares (snRNPs), o primeiro corte é feito pelo resíduo adenilil (no local da ramificação) através de um ataque nucleofílico no local dador 5′.
Imagem do centro: O terminal 5′ livre forma então uma ligação com o local da ramificação (fazendo a estrutura em laço).
Imagem de baixo: O segundo corte é feito no local 3′ do intrão e os exões são unidos.
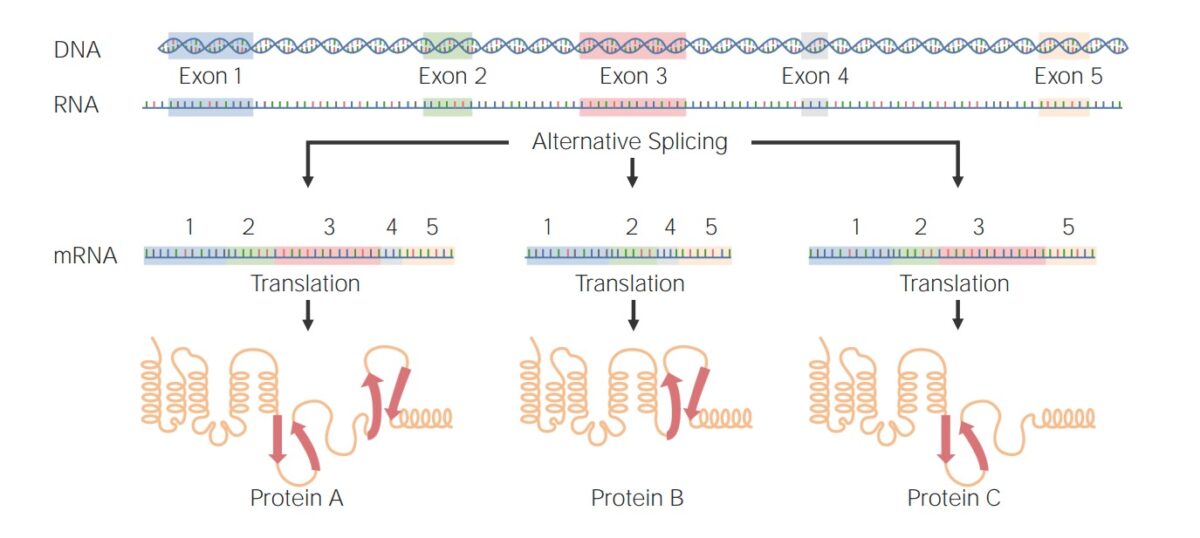
Exemplos de splicing alternativo:
Proteína A: Exões 1-5 foram unidos após splicing de intrões.
Proteínas B e C: Um exão foi seletivamente excluído para formar uma proteína diferente.
Geralmente, a sequência de DNA DNA A deoxyribonucleotide polymer that is the primary genetic material of all cells. Eukaryotic and prokaryotic organisms normally contain DNA in a double-stranded state, yet several important biological processes transiently involve single-stranded regions. DNA, which consists of a polysugar-phosphate backbone possessing projections of purines (adenine and guanine) and pyrimidines (thymine and cytosine), forms a double helix that is held together by hydrogen bonds between these purines and pyrimidines (adenine to thymine and guanine to cytosine). DNA Types and Structure é refletida no mRNA mRNA RNA sequences that serve as templates for protein synthesis. Bacterial mRNAs are generally primary transcripts in that they do not require post-transcriptional processing. Eukaryotic mRNA is synthesized in the nucleus and must be exported to the cytoplasm for translation. Most eukaryotic mRNAs have a sequence of polyadenylic acid at the 3′ end, referred to as the poly(a) tail. The function of this tail is not known for certain, but it may play a role in the export of mature mRNA from the nucleus as well as in helping stabilize some mRNA molecules by retarding their degradation in the cytoplasm. RNA Types and Structure maturado. A alteração da sequência ou edição do RNA RNA A polynucleotide consisting essentially of chains with a repeating backbone of phosphate and ribose units to which nitrogenous bases are attached. RNA is unique among biological macromolecules in that it can encode genetic information, serve as an abundant structural component of cells, and also possesses catalytic activity. RNA Types and Structure é uma exceção.
Edição “C-para-U”:
Edição “A-para-I”:
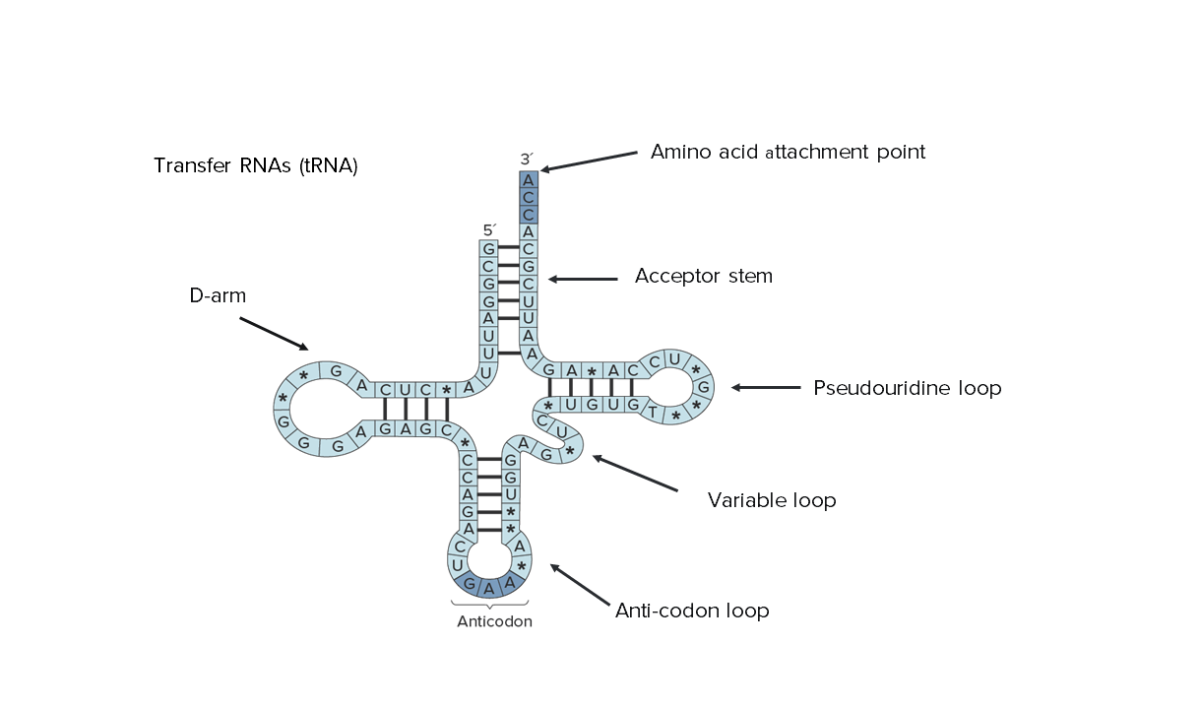
Estrutura secundária de RNA de transferência (tRNA). Note que toda a sua sequência pode ser vista, destacando o seu tamanho reduzido.
Imagem de Lecturio.