Embora a fidelidade do DNA DNA A deoxyribonucleotide polymer that is the primary genetic material of all cells. Eukaryotic and prokaryotic organisms normally contain DNA in a double-stranded state, yet several important biological processes transiently involve single-stranded regions. DNA, which consists of a polysugar-phosphate backbone possessing projections of purines (adenine and guanine) and pyrimidines (thymine and cytosine), forms a double helix that is held together by hydrogen bonds between these purines and pyrimidines (adenine to thymine and guanine to cytosine). DNA Types and Structure esteja altamente protegida, este pode ser danificado por vários fatores ambientais, espécies reativas de oxigénio e por erros na replicação. A reparação do DNA DNA A deoxyribonucleotide polymer that is the primary genetic material of all cells. Eukaryotic and prokaryotic organisms normally contain DNA in a double-stranded state, yet several important biological processes transiently involve single-stranded regions. DNA, which consists of a polysugar-phosphate backbone possessing projections of purines (adenine and guanine) and pyrimidines (thymine and cytosine), forms a double helix that is held together by hydrogen bonds between these purines and pyrimidines (adenine to thymine and guanine to cytosine). DNA Types and Structure é um processo contínuo em que a célula corrige os danos. A célula possui múltiplos mecanismos que pode usar para reparar o DNA DNA A deoxyribonucleotide polymer that is the primary genetic material of all cells. Eukaryotic and prokaryotic organisms normally contain DNA in a double-stranded state, yet several important biological processes transiently involve single-stranded regions. DNA, which consists of a polysugar-phosphate backbone possessing projections of purines (adenine and guanine) and pyrimidines (thymine and cytosine), forms a double helix that is held together by hydrogen bonds between these purines and pyrimidines (adenine to thymine and guanine to cytosine). DNA Types and Structure. Durante a replicação, a célula tem maquinaria de revisão dentro da própria DNA DNA A deoxyribonucleotide polymer that is the primary genetic material of all cells. Eukaryotic and prokaryotic organisms normally contain DNA in a double-stranded state, yet several important biological processes transiently involve single-stranded regions. DNA, which consists of a polysugar-phosphate backbone possessing projections of purines (adenine and guanine) and pyrimidines (thymine and cytosine), forms a double helix that is held together by hydrogen bonds between these purines and pyrimidines (adenine to thymine and guanine to cytosine). DNA Types and Structure polimerase. Para danos de DNA DNA A deoxyribonucleotide polymer that is the primary genetic material of all cells. Eukaryotic and prokaryotic organisms normally contain DNA in a double-stranded state, yet several important biological processes transiently involve single-stranded regions. DNA, which consists of a polysugar-phosphate backbone possessing projections of purines (adenine and guanine) and pyrimidines (thymine and cytosine), forms a double helix that is held together by hydrogen bonds between these purines and pyrimidines (adenine to thymine and guanine to cytosine). DNA Types and Structure de cadeia única, a célula pode usar técnicas de reparação por excisão e fotorreparação. Para as quebras de DNA DNA A deoxyribonucleotide polymer that is the primary genetic material of all cells. Eukaryotic and prokaryotic organisms normally contain DNA in a double-stranded state, yet several important biological processes transiently involve single-stranded regions. DNA, which consists of a polysugar-phosphate backbone possessing projections of purines (adenine and guanine) and pyrimidines (thymine and cytosine), forms a double helix that is held together by hydrogen bonds between these purines and pyrimidines (adenine to thymine and guanine to cytosine). DNA Types and Structure de cadeia dupla, a célula pode usar recombinação homóloga ou união não-homóloga das extremidades. Quando os processos normais de reparação do DNA DNA A deoxyribonucleotide polymer that is the primary genetic material of all cells. Eukaryotic and prokaryotic organisms normally contain DNA in a double-stranded state, yet several important biological processes transiently involve single-stranded regions. DNA, which consists of a polysugar-phosphate backbone possessing projections of purines (adenine and guanine) and pyrimidines (thymine and cytosine), forms a double helix that is held together by hydrogen bonds between these purines and pyrimidines (adenine to thymine and guanine to cytosine). DNA Types and Structure falham devido à idade, disfunção ou devido a um sistema sobrecarregado, os danos não reparados no DNA DNA A deoxyribonucleotide polymer that is the primary genetic material of all cells. Eukaryotic and prokaryotic organisms normally contain DNA in a double-stranded state, yet several important biological processes transiently involve single-stranded regions. DNA, which consists of a polysugar-phosphate backbone possessing projections of purines (adenine and guanine) and pyrimidines (thymine and cytosine), forms a double helix that is held together by hydrogen bonds between these purines and pyrimidines (adenine to thymine and guanine to cytosine). DNA Types and Structure podem levar à apoptose, senescência celular ou tumores malignos.
Last updated: Dec 15, 2025
Os danos no DNA DNA A deoxyribonucleotide polymer that is the primary genetic material of all cells. Eukaryotic and prokaryotic organisms normally contain DNA in a double-stranded state, yet several important biological processes transiently involve single-stranded regions. DNA, which consists of a polysugar-phosphate backbone possessing projections of purines (adenine and guanine) and pyrimidines (thymine and cytosine), forms a double helix that is held together by hydrogen bonds between these purines and pyrimidines (adenine to thymine and guanine to cytosine). DNA Types and Structure podem ser causados por:
A revisão ocorre durante a replicação de ADN. A DNA DNA A deoxyribonucleotide polymer that is the primary genetic material of all cells. Eukaryotic and prokaryotic organisms normally contain DNA in a double-stranded state, yet several important biological processes transiently involve single-stranded regions. DNA, which consists of a polysugar-phosphate backbone possessing projections of purines (adenine and guanine) and pyrimidines (thymine and cytosine), forms a double helix that is held together by hydrogen bonds between these purines and pyrimidines (adenine to thymine and guanine to cytosine). DNA Types and Structure polimerase (o complexo enzimático que replica o DNA DNA A deoxyribonucleotide polymer that is the primary genetic material of all cells. Eukaryotic and prokaryotic organisms normally contain DNA in a double-stranded state, yet several important biological processes transiently involve single-stranded regions. DNA, which consists of a polysugar-phosphate backbone possessing projections of purines (adenine and guanine) and pyrimidines (thymine and cytosine), forms a double helix that is held together by hydrogen bonds between these purines and pyrimidines (adenine to thymine and guanine to cytosine). DNA Types and Structure):
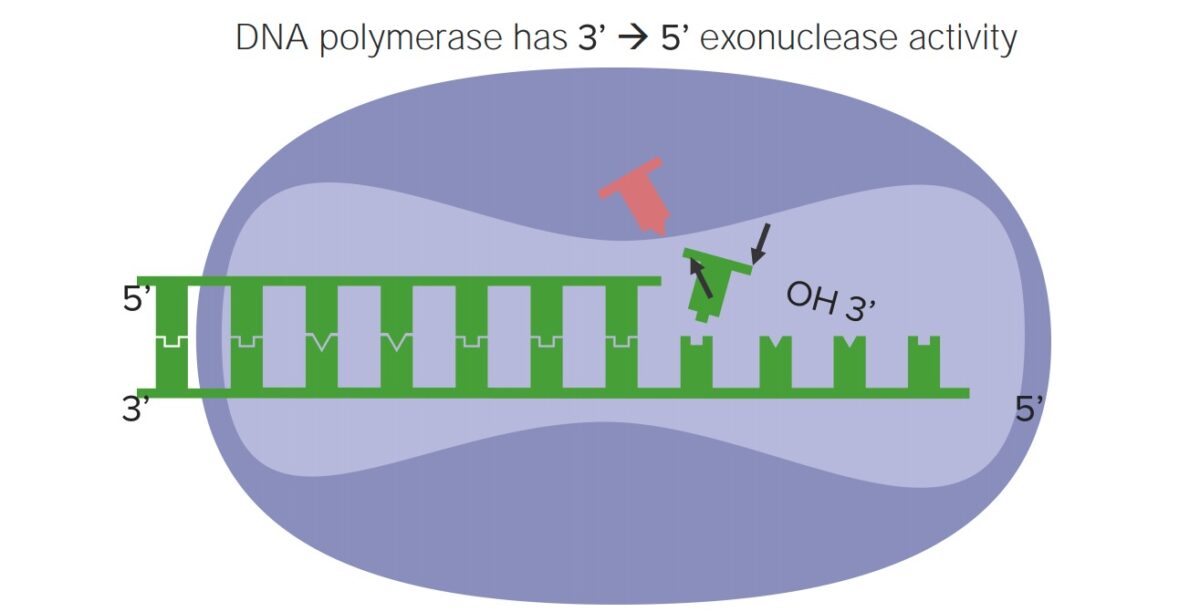
Atividade de revisão da DNA polimerase
Image by Lecturio.A radiação UV provoca a formação de dímeros de pirimidina (T ou C) através de ligações covalentes entre bases adjacentes, criando uma alteração conformacional (“bulge”) no DNA DNA A deoxyribonucleotide polymer that is the primary genetic material of all cells. Eukaryotic and prokaryotic organisms normally contain DNA in a double-stranded state, yet several important biological processes transiently involve single-stranded regions. DNA, which consists of a polysugar-phosphate backbone possessing projections of purines (adenine and guanine) and pyrimidines (thymine and cytosine), forms a double helix that is held together by hydrogen bonds between these purines and pyrimidines (adenine to thymine and guanine to cytosine). DNA Types and Structure. Estes defeitos podem ser reparados através de um processo chamado fotorreparação, ou fotorreativação.
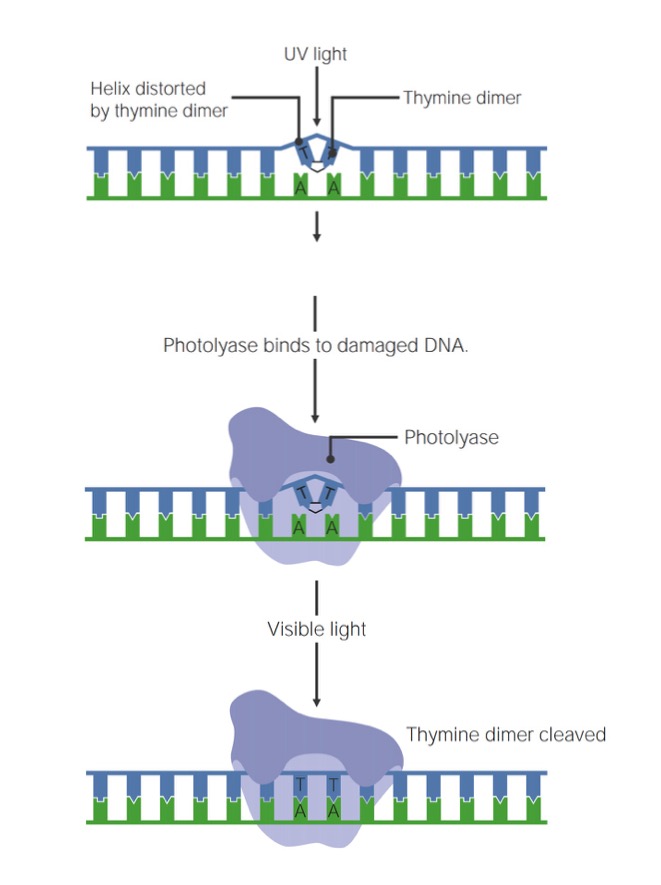
Passos da fotorreparação
Image by Lecturio.A célula tem 3 mecanismos primários para reparar os danos feitos a uma cadeia simples de DNA DNA A deoxyribonucleotide polymer that is the primary genetic material of all cells. Eukaryotic and prokaryotic organisms normally contain DNA in a double-stranded state, yet several important biological processes transiently involve single-stranded regions. DNA, which consists of a polysugar-phosphate backbone possessing projections of purines (adenine and guanine) and pyrimidines (thymine and cytosine), forms a double helix that is held together by hydrogen bonds between these purines and pyrimidines (adenine to thymine and guanine to cytosine). DNA Types and Structure:
Todos os 3 mecanismos seguem o mesmo processo geral:
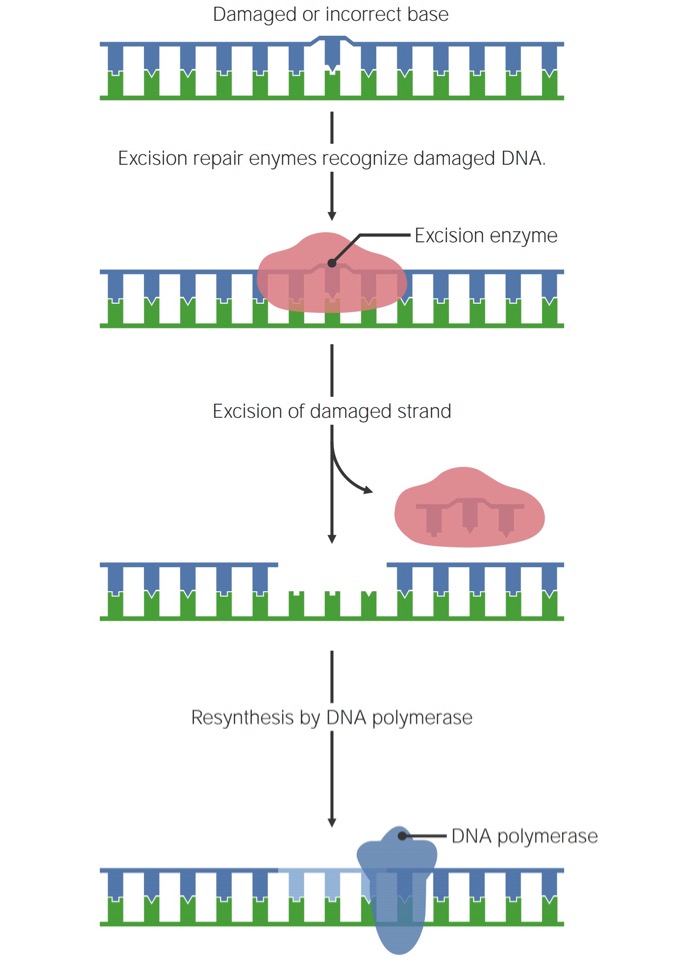
Mecanismo geral de reparação de DNA de cadeia simples
Image by Lecturio.Na REB REB Research Ethics Boards (REB) are bodies within institutions (i.e. universities, hospitals) that assess the ethical soundness of a study to ultimately indicate if the study can be carried out without ethical liability. Research Ethics, uma única base danificada é excisada e substituída.
Em geral, é mais MAIS Androgen Insensitivity Syndrome difícil reparar danos em DNA DNA A deoxyribonucleotide polymer that is the primary genetic material of all cells. Eukaryotic and prokaryotic organisms normally contain DNA in a double-stranded state, yet several important biological processes transiently involve single-stranded regions. DNA, which consists of a polysugar-phosphate backbone possessing projections of purines (adenine and guanine) and pyrimidines (thymine and cytosine), forms a double helix that is held together by hydrogen bonds between these purines and pyrimidines (adenine to thymine and guanine to cytosine). DNA Types and Structure de cadeia dupla porque não há nenhuma cadeia modelo em que se basear. Os 2 mecanismos primários para fixar quebras de DNA DNA A deoxyribonucleotide polymer that is the primary genetic material of all cells. Eukaryotic and prokaryotic organisms normally contain DNA in a double-stranded state, yet several important biological processes transiently involve single-stranded regions. DNA, which consists of a polysugar-phosphate backbone possessing projections of purines (adenine and guanine) and pyrimidines (thymine and cytosine), forms a double helix that is held together by hydrogen bonds between these purines and pyrimidines (adenine to thymine and guanine to cytosine). DNA Types and Structure de cadeia dupla são a recombinação homóloga e a união de extremidades não homóloga.
Na recombinação homóloga (RH), é usado como um modelo o quase idêntico cromatídeo irmão ou o cromossoma homólogo
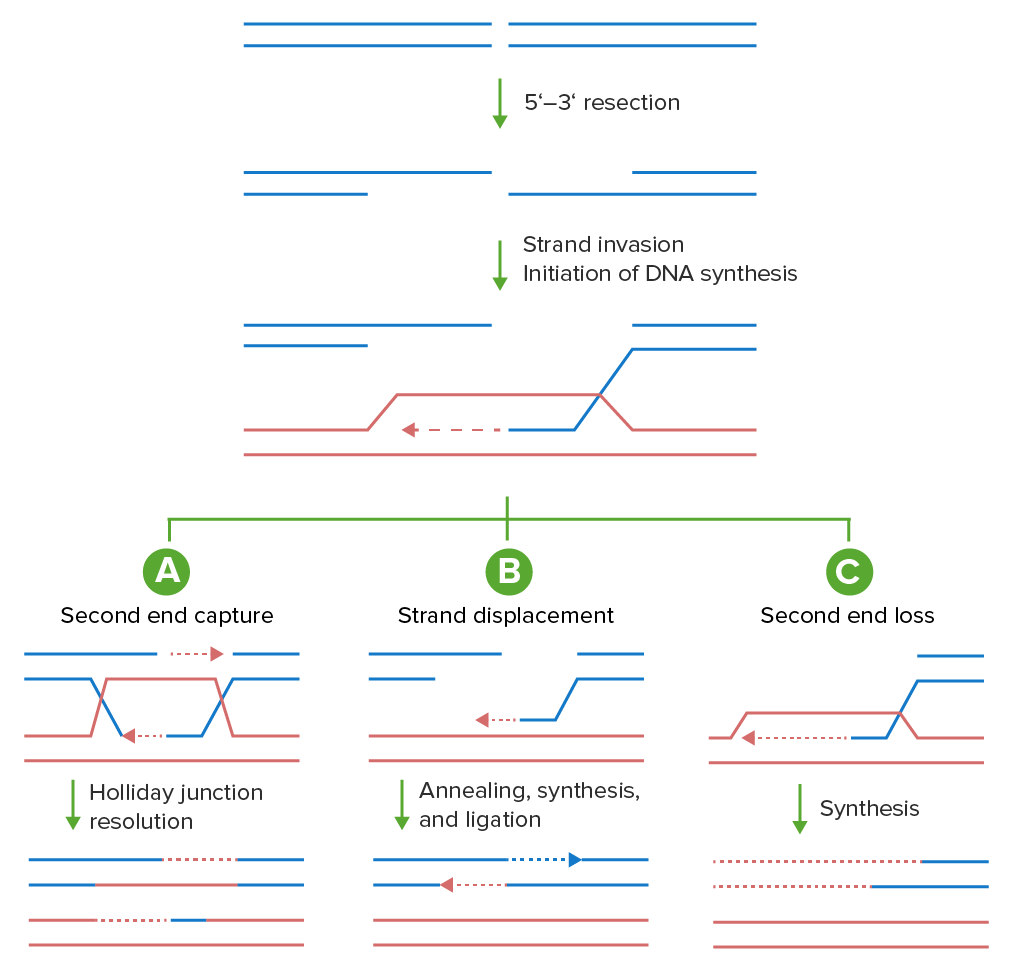
Modelos de recombinação homóloga:
As quebras de cadeia dupla podem ser reparadas utilizando a maquinaria de recombinação homóloga de diversas formas. As extremidades de DNA são primeiro processadas em caudas de DNA 3′ de cadeia única. Estas caudas invadem um modelo homólogo (vermelho), servindo de primer para uma nova síntese de DNA (linha tracejada). Estão representados 3 resultados possíveis desta invasão.
A: No reparo canónico de quebras de cadeia dupla (DSBR pela sigla em inglês), tanto a cadeia invasora inicial como a extremidade capturada juntam-se ao modelo homólogo e servem como primer para nova síntese de DNA, resultando numa dupla junção de Holliday que pode ser resolvida por nucleases num produto cruzado ou não cruzado (na figura está representado o produto não cruzado).
B: Alternativamente, após a cauda de DNA cadeia simples invadir o modelo homólogo, é preparada uma rodada de síntese de DNA a partir da extremidade 3′ (linha tracejada vermelha). O anelamento dependente da síntese da cadeia (SDSA) ocorre quando a cadeia invasora, juntamente com o segmento recém-sintetizado, é desenrolada por uma hélice e recozida com a outra extremidade retirada.
C: Na replicação induzida por falha (BIR), 1 extremidade da quebra de cadeia dupla é perdida e extremidade restante invade a síntese de DNA do modelo homólogo, servindo de primer para a síntese até ao final do cromossoma.