A tradução é o processo de sintetizar uma proteína a partir de um transcrito de RNA mensageiro (mRNA). Este processo é dividido em três fases primárias: iniciação, alongamento e finalização. A tradução é catalisada por estruturas conhecidas como ribossomas, grandes complexos de proteínas e RNA ribossomal (rRNA). O ribossoma "lê" o mRNA, e traz os RNA de transferência (tRNAs), cada um ligado a um aminoácido específico, na ordem correta. Estes aminoácidos são então ligados uns aos outros por um componente enzimático do ribossoma conhecido como peptidil transferase. A tradução pode ser regulada em várias etapas, inclusive através de um processo conhecido como interferência de RNA. Este processo envolve pequenos segmentos de RNA de cadeia dupla que são capazes de inibir a tradução de mRNA.
Última atualização: Aug 15, 2022
O código genético é como os organismos traduzem uma sequência de bases numa proteína real.
Os mRNA são traduzidos em proteínas pelos ribossomas e pelos RNA de transferência.
Os tRNA carregam os aminoácidos até aos ribossomas, onde eles se ligam ao mRNA, alinhando os aminoácidos que se ligarão para formar o polipéptido em crescimento.
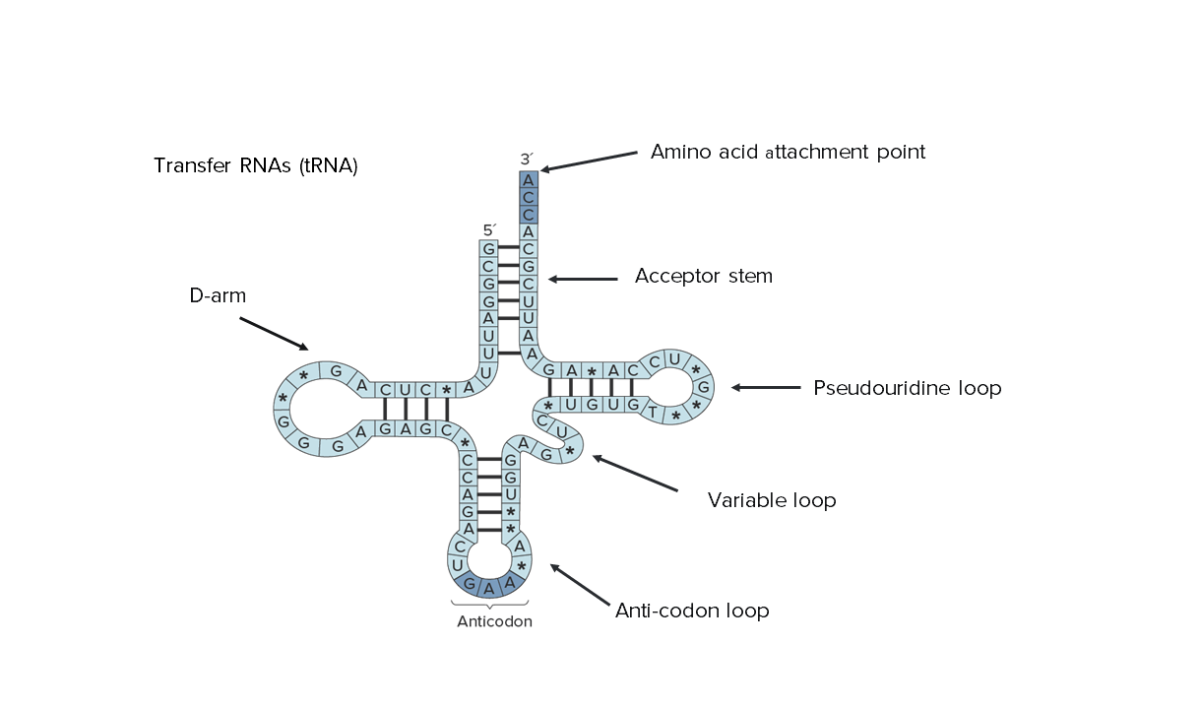
Estrutura secundária do RNA transferência (tRNA). Note-se que toda a sua sequência pode ser vista, indicando o tamanho reduzido.
Image by Lecturio.
Carregamento de um tRNA na aminoacil sintase
Image by Lecturio.Os ribossomas são complexos catalíticos que incluem componentes proteicos e rRNA. Dentro do complexo ribossómico, o mRNA é lido por tRNAs e é criado um polipéptido.
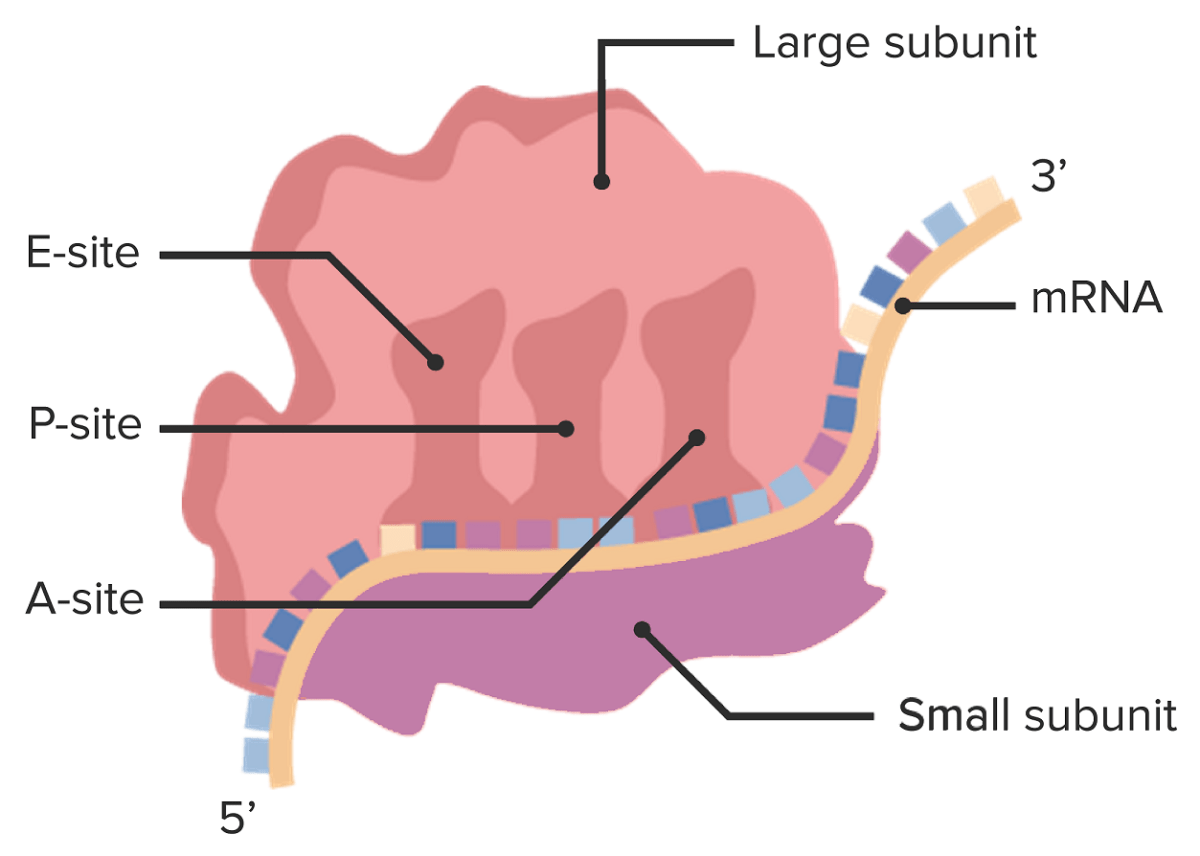
Estrutura do ribossoma que demonstra a grande subunidade no topo, com os locais de ligação A, P, e E para tRNAs carregados. A subunidade menor está abaixo do mRNA.
Image by Lecturio.| Procariontes | Eucariontes | |
|---|---|---|
| Tamanho da subunidade pequena | 30 S | 40 S |
| Tamanho da subunidade grande | 50 S | 60 S |
| Número de proteínas | 52 | 88 |
| Número de rRNA | 3 | 4 |
| Tamanho dos rRNA homólogos na subunidade pequena | 16 S | 18 S |
| Tamanhos de rRNA homólogos na subunidade grande |
|
|
| Tamanho do rRNA na subunidade grande sem um homólogo procariótico | 5,8 S |
O início da tradução envolve a montagem do ribossoma no mRNA na direção correta e encontrar o codão inicial.
Assim que a subunidade pequena se liga à extremidade 5′ do mRNA, esta começa a procurar o local de início.
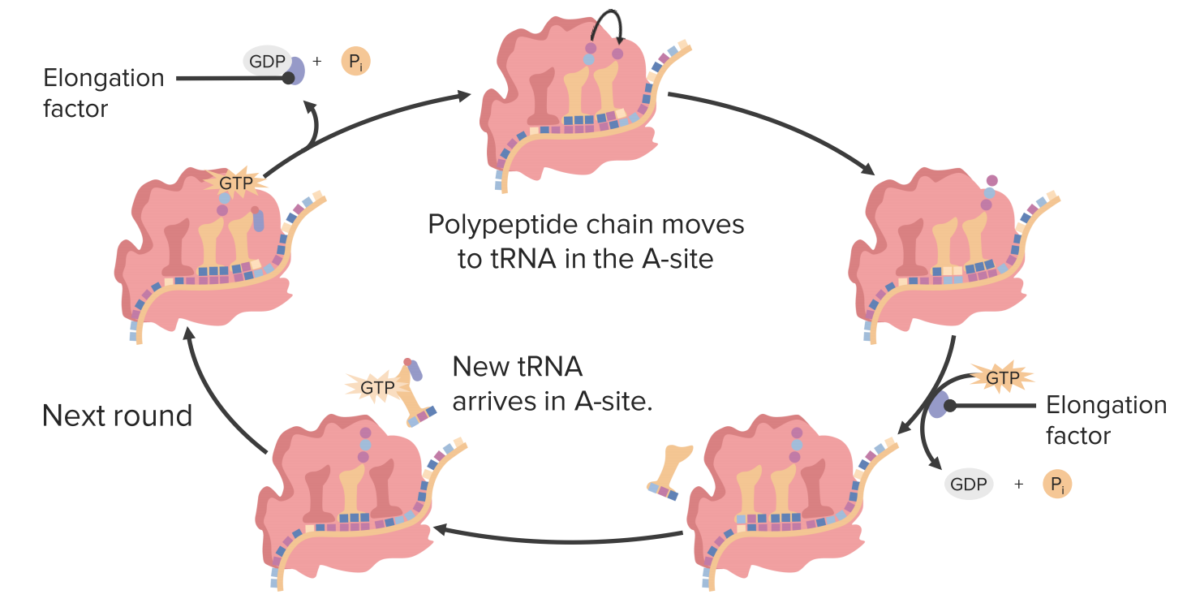
O ciclo de alongamento
Image by Lecturio.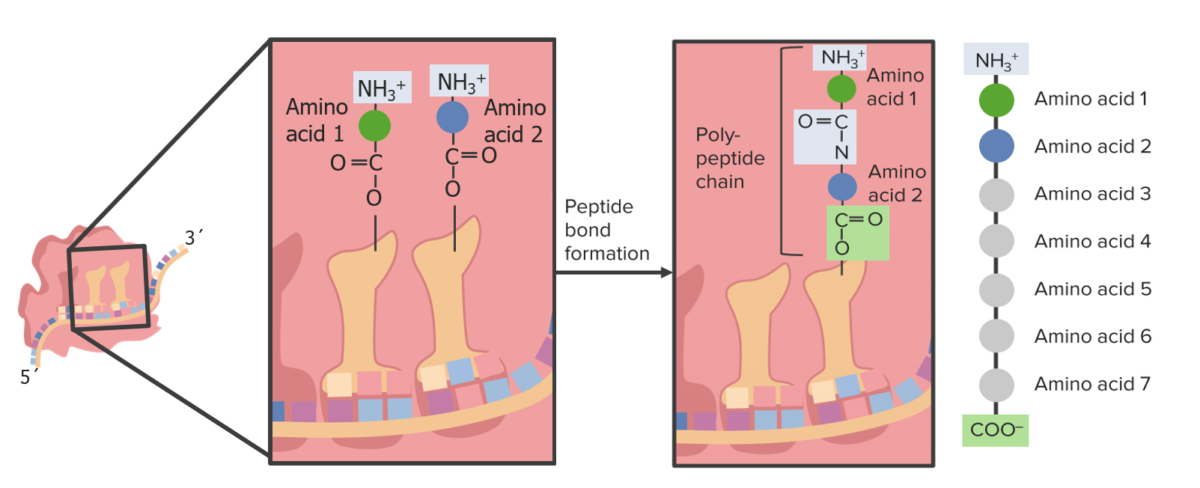
Forma-se uma ligação peptídica dentro do ribossoma.
Image by Lecturio.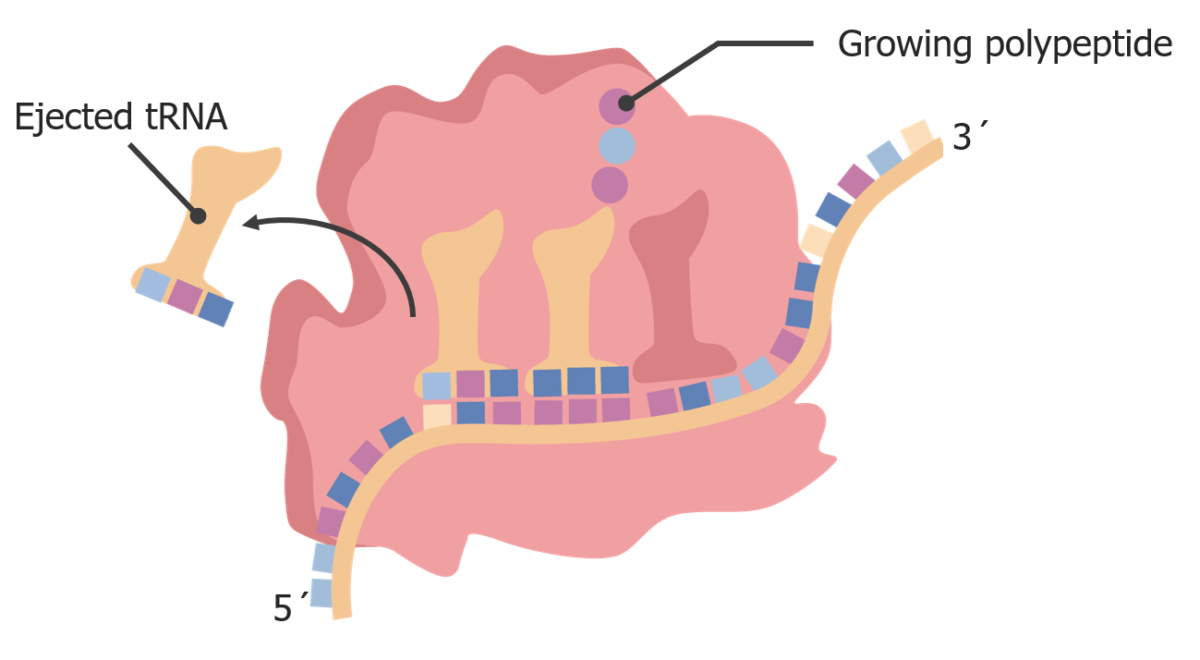
Quando o ribossoma se desloca, o tRNA “vazio” move-se para a posição E sendo ejetado do ribossoma.
Image by Lecturio.A terminação ocorre quando o ribossoma atinge um codão de paragem.
A tradução pode ser regulada no nível de iniciação, do alongamento ou da terminação, principalmente através de “up-regulation” (regulação positiva, a favorecendo a síntese) e “down-regulation” (regulação negativa, contra a síntese) de fatores de iniciação, alongamento e terminação. A tradução é também regulada através RNA de interferência, splicing alternativo e edição de RNA.
RNA de interferência (RNAi) são pequenas moléculas de RNA que causam interferência na tradução, que acabam por inibir a tradução de mRNA específicos.
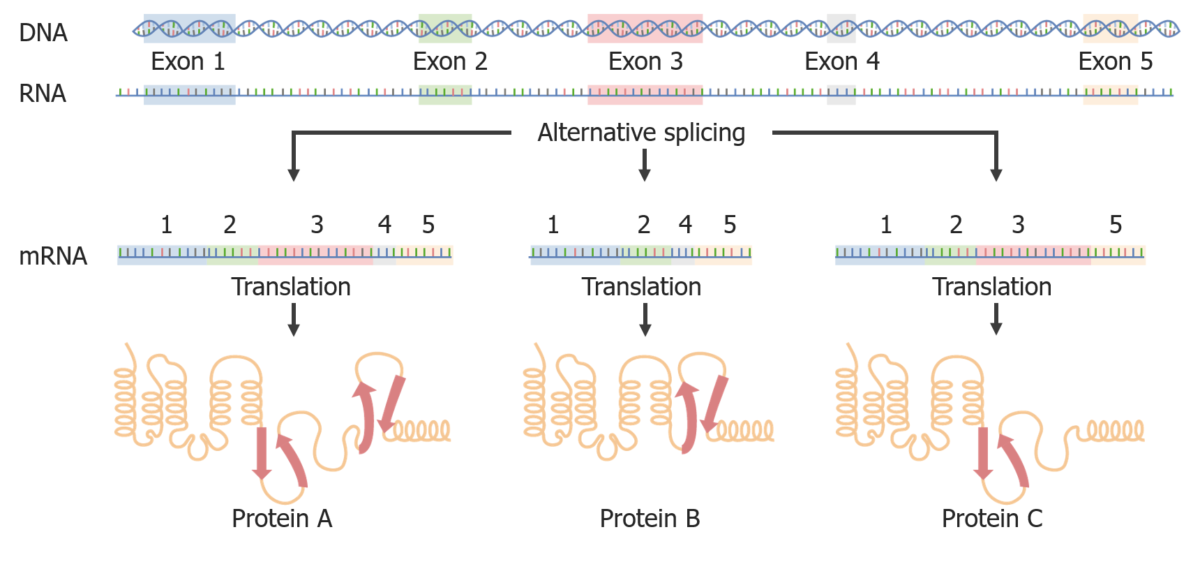
Splicing alternativo:
Através do splicing de várias maneiras, diferentes proteínas podem ser criadas a partir do mesmo mRNA.