El procesamiento postraduccional de las proteínas (incluida la modificación postraduccional) es el plegado, clasificación, escisión y las modificaciones necesarias para que una proteína sea funcional después de ser traducida. A medida que la proteína se pliega, forma complejas estructuras secundarias, terciarias y cuaternarias. Además, se pueden añadir nuevos grupos funcionales o moléculas a la cadena polipeptídica, incluyendo grupos fosforilo, metilo o acetilo; carbohidratos y lípidos. Las proteínas también tienen que ser clasificadas en el compartimento intracelular correcto para llevar a cabo su función, ser empaquetadas para su secreción o ser insertadas en la membrana celular adecuada.
Última actualización: Abr 17, 2025
Los aminoácidos son los componentes básicos de las proteínas. Entender los fundamentos de los aminoácidos permite comprender mejor el plegamiento y la modificación de las proteínas.
Los aminoácidos que componen las proteínas se conocen como α-aminoácidos. Cada uno de estos aminoácidos tiene un carbono central conocido como “carbono alfa”, que realiza 4 enlaces:
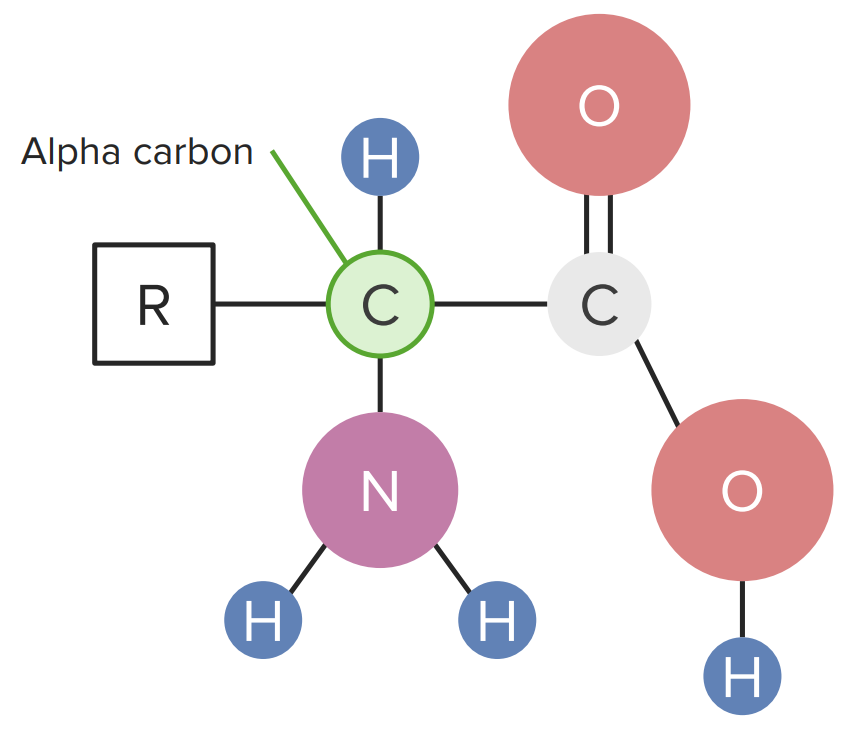
Diagrama de un aminoácido
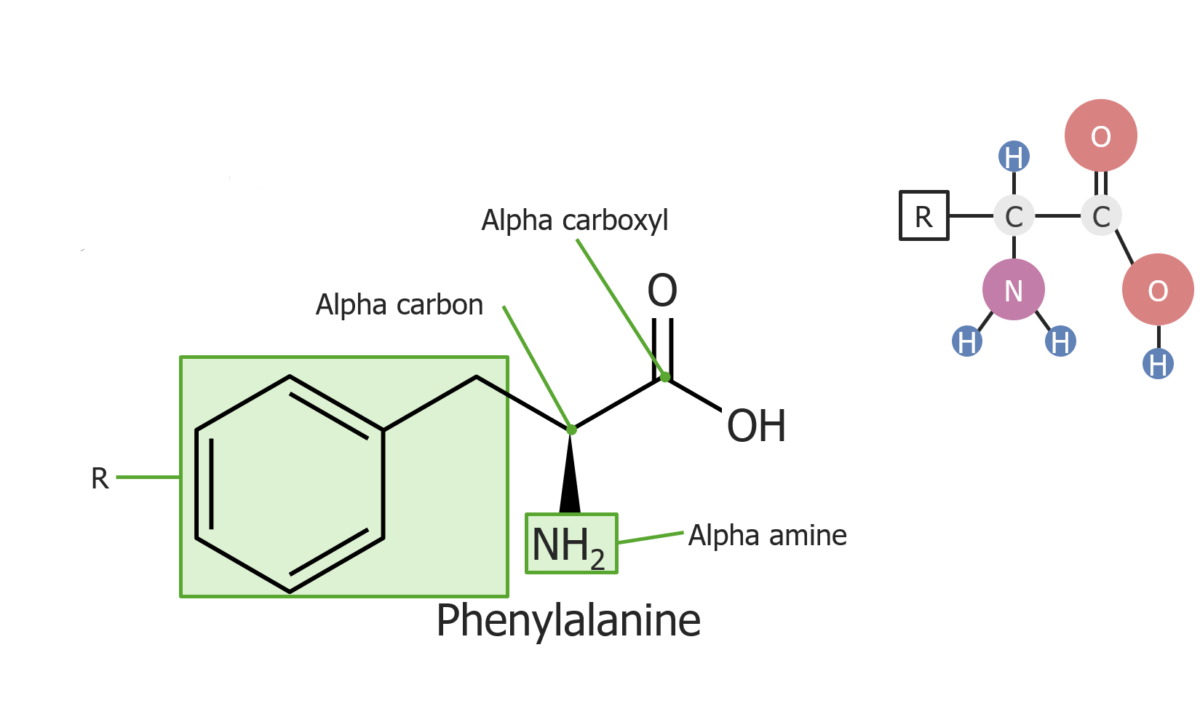
Ejemplo del aminoácido fenilalanina
Los aminoácidos pueden clasificarse por las características de sus grupos R, que pueden ser:

Clasificación de los aminoácidos por grupo R
Imagen por Lecturio.La estructura de las proteínas, que a menudo se denomina plegado de proteínas, tiene 4 niveles. Estos niveles son:
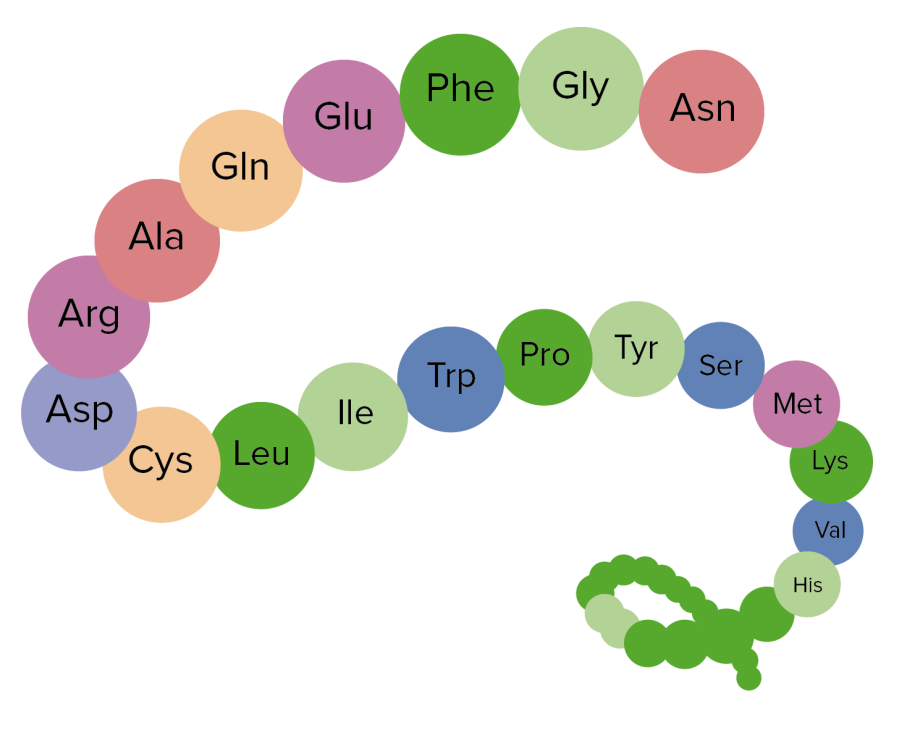
Ejemplo de estructura primaria de una proteína
Imagen por Lecturio.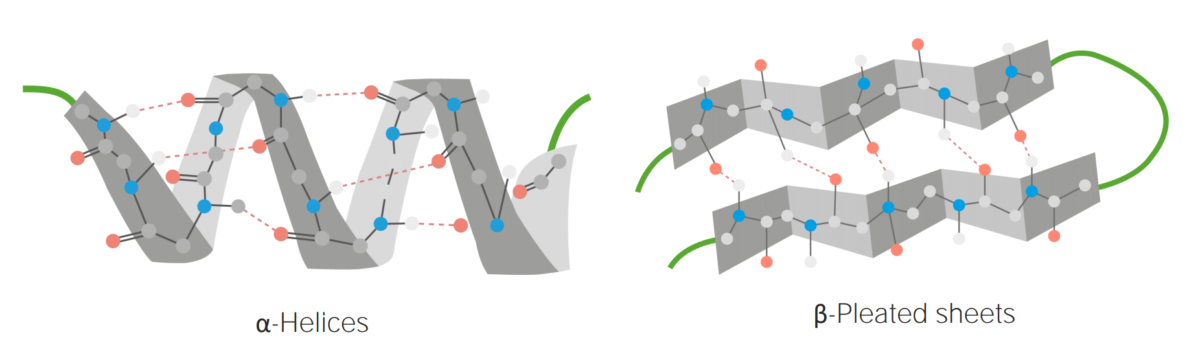
Ejemplos de α-hélices y láminas β-plegadas
Imagen por Lecturio.La estructura terciaria es el complejo bucle y plegado que se produce como resultado de las interacciones y los enlaces entre las porciones de la proteína que están más separadas. Algunos ejemplos de interacciones que crean una estructura terciaria son:
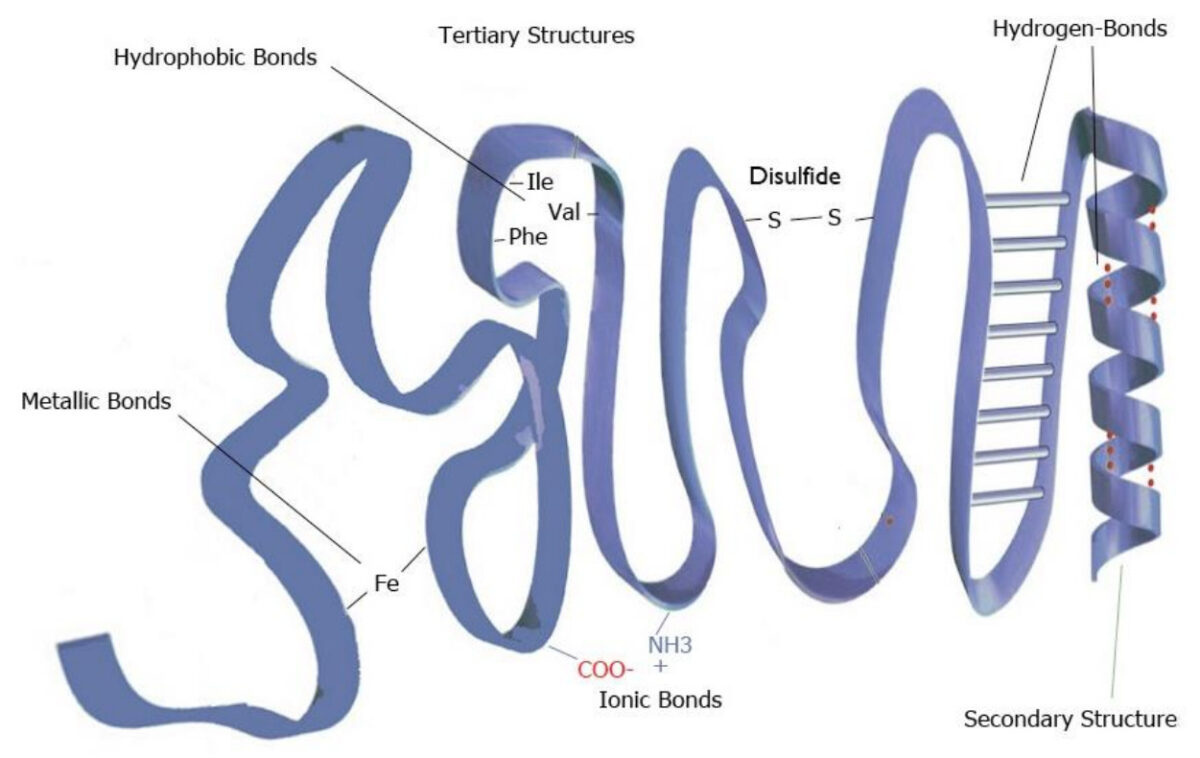
Ejemplo de estructura terciaria
Imagen por Lecturio.En una estructura cuaternaria, varias subunidades de una proteína se unen para formar una única proteína.
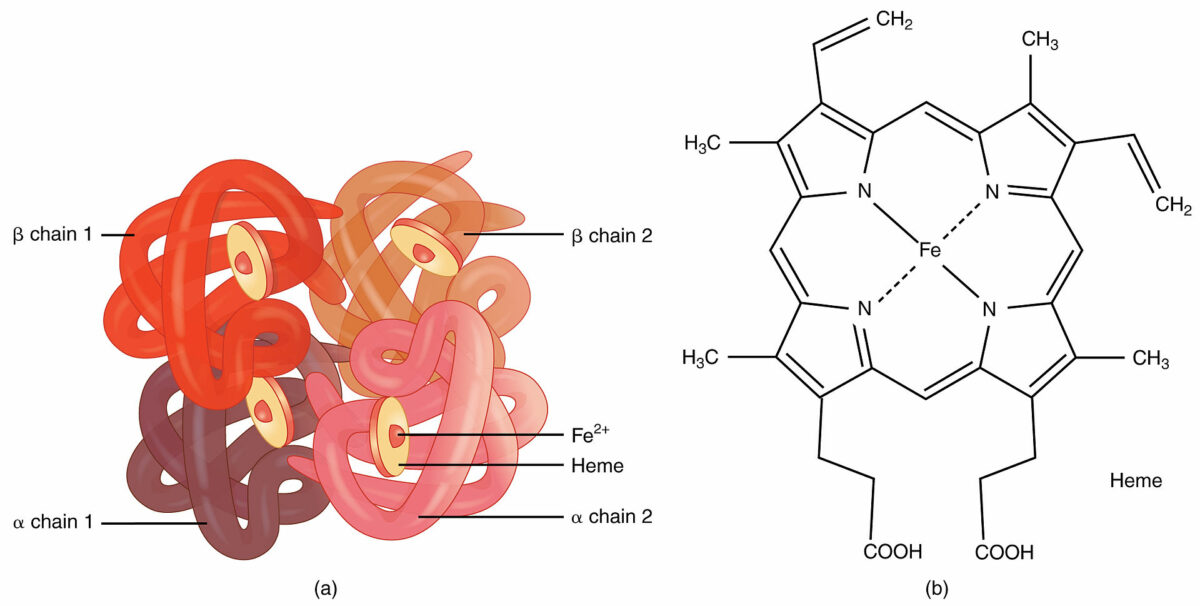
Hemoglobina:
Un ejemplo de estructura cuaternaria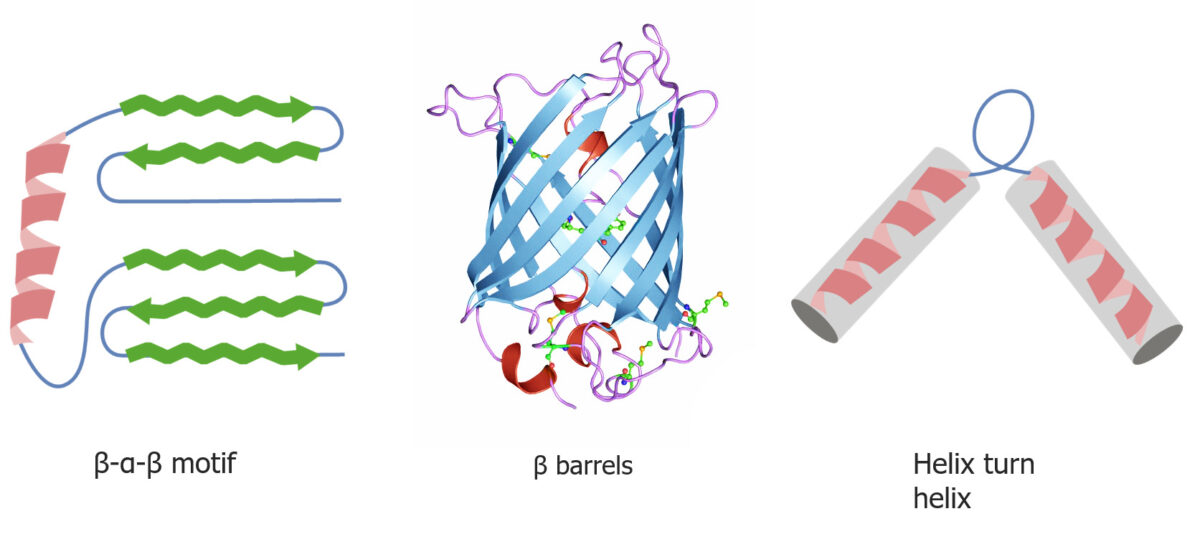
Motivos de plegado cuaternario y terciario de las proteínas
Las proteínas chaperonas ayudan al plegado de las proteínas.
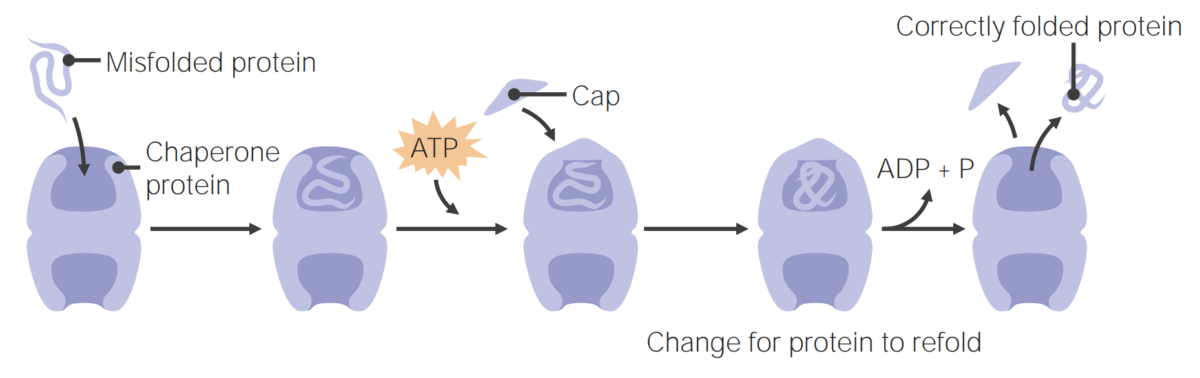
Las proteínas chaperonas ayudan al plegado de las proteínas
Imagen por Lecturio.Una proteína desnaturalizada es una proteína que se ha desdoblado y ya no es funcional. Este desdoblamiento se produce bajo ciertas condiciones, que incluyen cambios en:
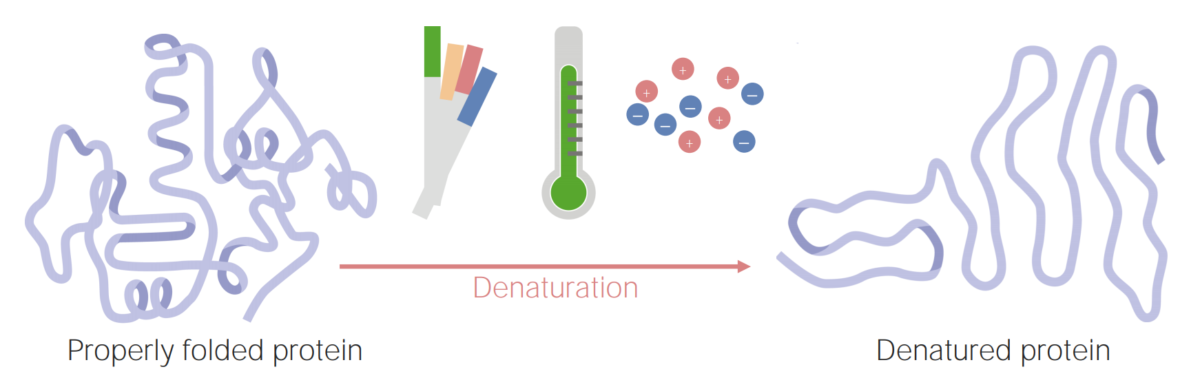
Las proteínas pueden desnaturalizarse (o desdoblarse) como resultado de cambios en el pH, temperatura o concentración iónica.
Imagen por Lecturio.Las proteínas deben ser clasificadas y acabarán permaneciendo en la célula, colocándose en la pared celular o siendo exportadas/secretadas.
Las proteínas destinadas a la superficie celular y/o a la secreción de la célula se sintetizan en el retículo endoplásmico rugoso:
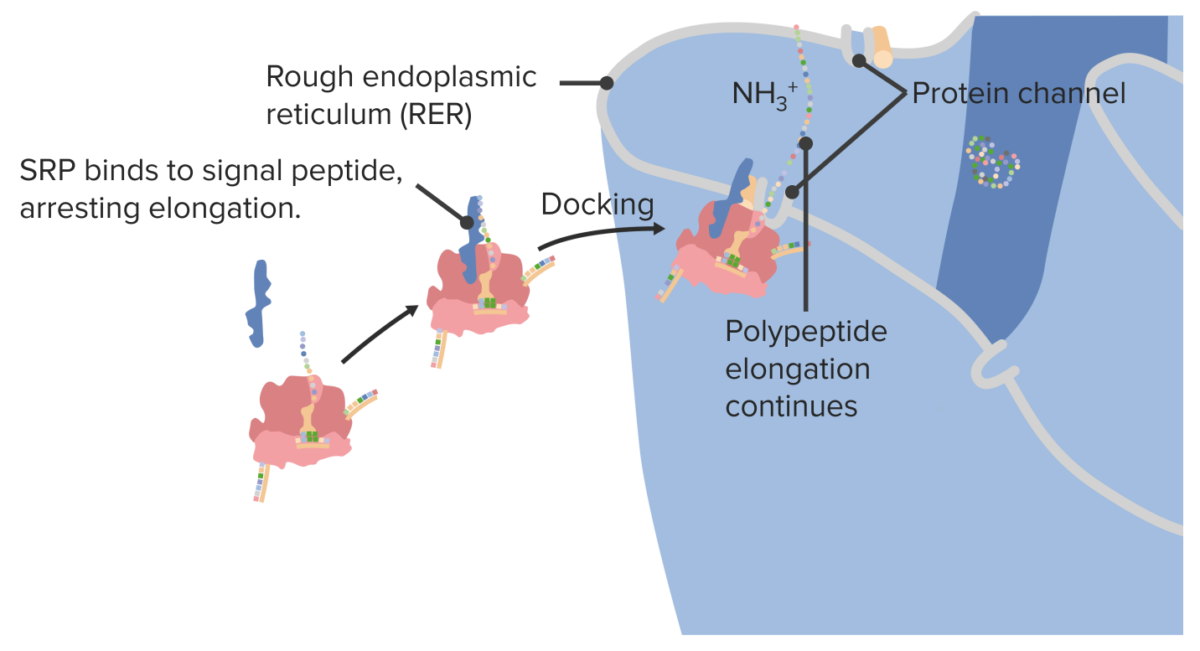
Acoplamiento de un ribosoma en el retículo endoplásmico rugoso
SRP: proteína de reconocimiento de señales
Después de sintetizar un polipéptido, se somete a una nueva modificación para formar una proteína funcional. Esta modificación puede incluir el corte de partes de la cadena polipeptídica o la adición de un grupo funcional.
La escisión de proteínas es el proceso de eliminación de ciertos polipéptidos para que la proteína sea funcional.
Las proteínas se modifican además mediante la adición covalente de grupos funcionales y otras moléculas.
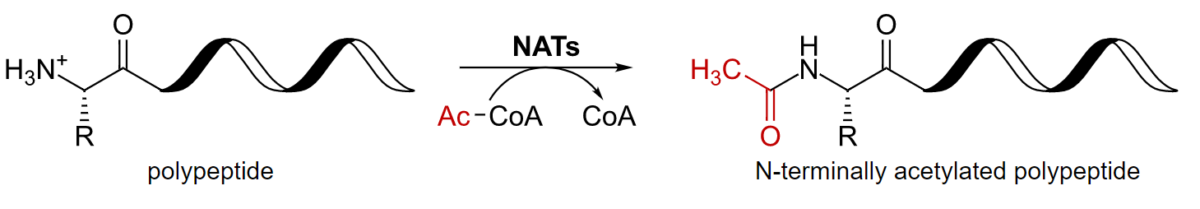
Acetilación de un polipéptido
CoA: coenzima A
NAT: acetiltransferasas N-terminales
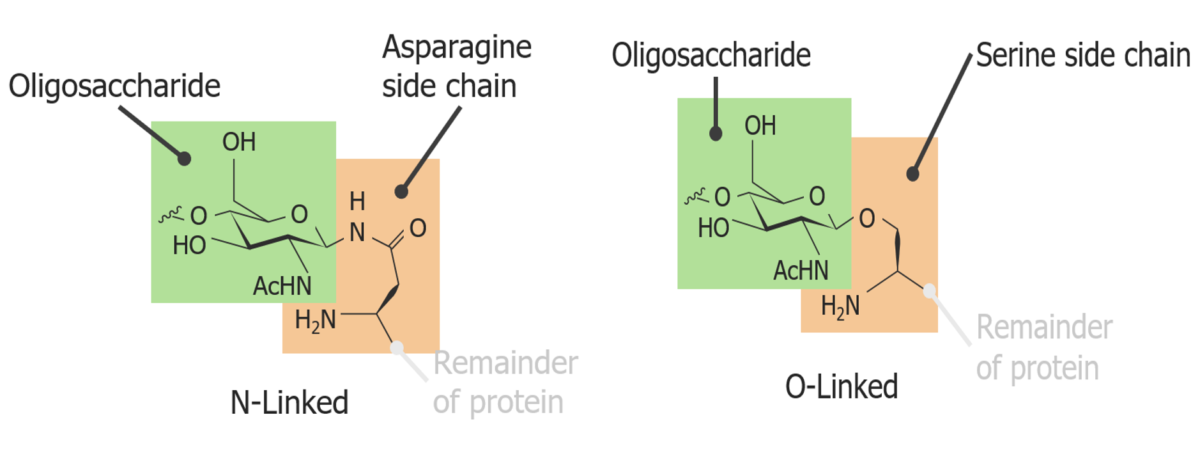
Una glicoproteína ligada a la N frente a una ligada a la O
Imagen por Lecturio.Las anomalías en la modificación postraduccional y/o en el plegamiento o la clasificación de las proteínas pueden dar lugar a una serie de afecciones clínicamente importantes.