Las purinas y las pirimidinas son compuestos aromáticos heterocíclicos que, junto con los grupos azúcar y fosfato, forman los componentes importantes de los nucleótidos. Las purinas incluyen adenina y guanina, mientras que las pirimidinas incluyen timina (en el ADN), uracilo (en el ARN) y citosina. La síntesis de nucleótidos de purina sigue una serie de reacciones que utilizan donantes de carbono, aminoácidos (e.g., glutamina, aspartato) y bicarbonato. La vía de novo genera inosina monofosfato (IMP), que es el precursor de adenosina monofosfato (AMP) y guanosina monofosfato (GMP). La síntesis de purinas se regula en los 1ros 2 pasos. La síntesis de nucleótidos de pirimidina también sigue diferentes reacciones, produciendo uridina monofosfato (UMP), que se convierte en uridina trifosfato (UTP) y citidina trifosfato (CTP, por sus siglas en inglés). Para la timina, una parte de los desoxirribonucleótidos, se requiere ribonucleósido reductasa para reducir el resto de ribosa. La degradación de los nucleótidos da como resultado la producción de xantina y luego de ácido úrico en purinas, mientras que las pirimidinas producen los aminoácidos, β-alanina y β-aminobutirato.
Última actualización: Sep 20, 2023
Bases nitrogenadas:
Nucleósidos: 2 componentes:
Un enlace beta-N-glucosídico une el 1er carbono del azúcar pentosa y N9 de una purina o N1 de una pirimidina (e.g., adenosina, guanosina, citidina, timidina, uridina, inosina).
Nucleótidos: 3 componentes principales:
Estas moléculas forman el esqueleto del ADN (e.g., adenosina monofosfato, guanosina monofosfato, citidina monofosfato)
> 1 grupo fosfato:
La esterificación de los grupos fosfato forma los correspondientes nucleósidos difosfatos y trifosfatos (e.g., adenosina trifosfato (ATP), adenosina difosfato (ADP)).
Ácido nucleico:
Polímero de nucleótidos (e.g., ácido ribonucleico (ARN)).
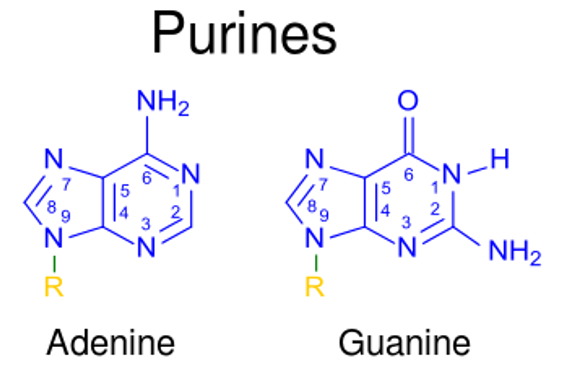
Estructura de las purinas adenina y guanina
Imagen: “123” por Kevin Ahern. Licencia: Dominio Público, recortada por Lecturio.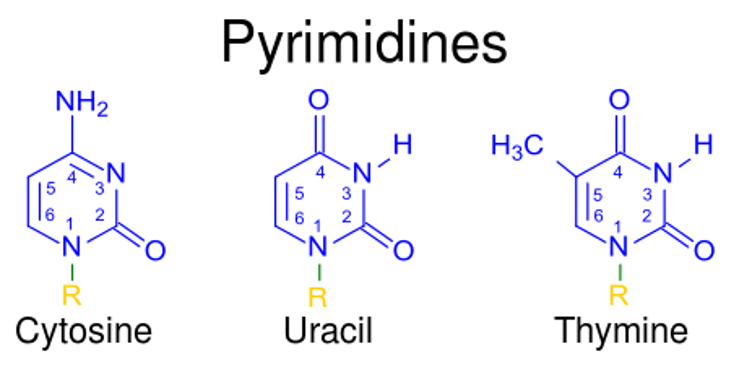
Nucleótidos de pirimidina y sus estructuras.
Imagen: “123” por Kevin Ahern. Licencia: Dominio Público, recortada por Lecturio.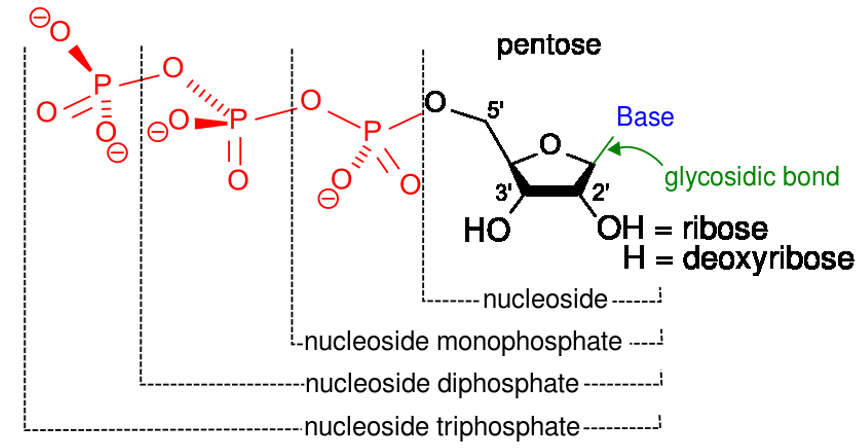
Componentes que forman un nucleótido
Imagen: “123” por Kevin Ahern. Licencia: Dominio Público, recortada por Lecturio.Las principales funciones de los nucleótidos:
Síntesis de 5-fosforribosil-1-pirofosfato
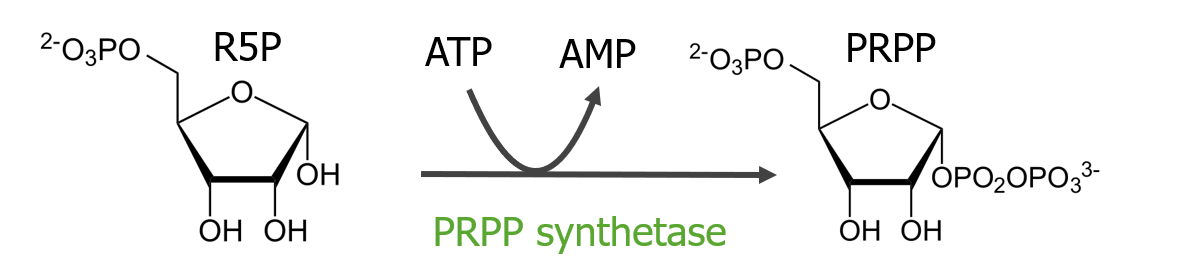
Síntesis de fosforribosil pirofosfato (PRPP):
La ribosa-5-fosfato (R5P) se convierte en PRPP. Los fosfatos provienen del ATP y se produce AMP. La enzima para la conversión es la PRPP sintetasa.
Formación de 5-fosforribosilamina
Conversión de 5-fosforribosilamina en ribonucleótido de glicinamida
Formilación del ribonucleótido de glicinamida a ribonucleótido de formilglicinamida
Conversión del ribonucleótido de formilglicinamida a ribonucleótido de formilglicinamidina
Formación del anillo de purina imidazol
Carboxilación del ribonucleótido de 5-aminoimidazol
Formación del ribonucleótido de 5-aminoimidazol-4-(N-succinilcarboxamida)
Eliminación de fumarato
Formilación para formar el ribonucleótido de 5-formaminoimidazol-4-carboxamida
Ciclación para formar IMP
| Paso | Reacción | Átomo añadido | Enzima | Producto |
|---|---|---|---|---|
| 1 | Ribosa-5-fosfato → PRPP | Fosfatos (del ATP) | PRPP sintetasa | PRPP |
| 2 | PRPP + glutamina → 5-fosforribosilamina | N9 (de la glutamina) | Amidofosforribosiltransferasa | PRA |
| 3 | Conversión de PRA a GAR | C4, C5, N7 (de la glicina) | GAR sintetasa | GAR |
| 4 | Formilación de GAR a FGAR | C8 (del THF de formilo) | GAR transformilasa | FGAR |
| 5 | Conversión de FGAR a FGAM | N3 (de la glutamina) | FGAM sintetasa | FGAM |
| 6 | Cierre del anillo, formando AIR | AIR sintetasa | AIR | |
| 7 | Carboxilación del AIR | C6 (del bicarbonato) | AIR carboxilasa | AICAR |
| 8 | Formación de SAICAR | N1 (del aspartato) | SAICAR sintetasa | SAICAR |
| 9 | Eliminación de fumarato, formación de AICAR | Adenilosuccinato liasa | AICAR | |
| 10 | FAICAR formado | C2 (del THF de formilo) | AICAR transformilasa | FAICAR |
| 11 | IMP formado | IMP ciclohidrolasa | IMP |
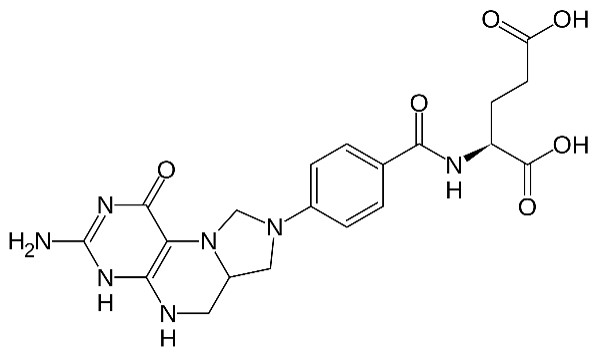
Estructura del folato
Imagen por Lecturio.La inosina monofosfato se convierte en adenina y guanina como AMP y GMP. Formado a partir de GMP, la guanosina trifosfato (GTP) proporciona la energía para convertir IMP en AMP.
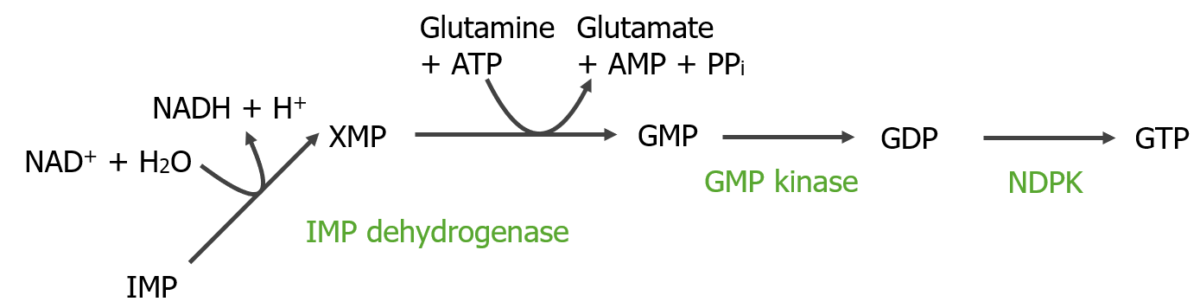
Conversión de IMP a GMP y luego a GTP:
NAD+: nicotinamida adenina dinucleótido (oxidado)
NADH: nicotinamida adenina dinucleótido (reducido)
NDPK: nucleósido difosfato quinasa (por sus siglas en inglés)
PPi: pirofosfato
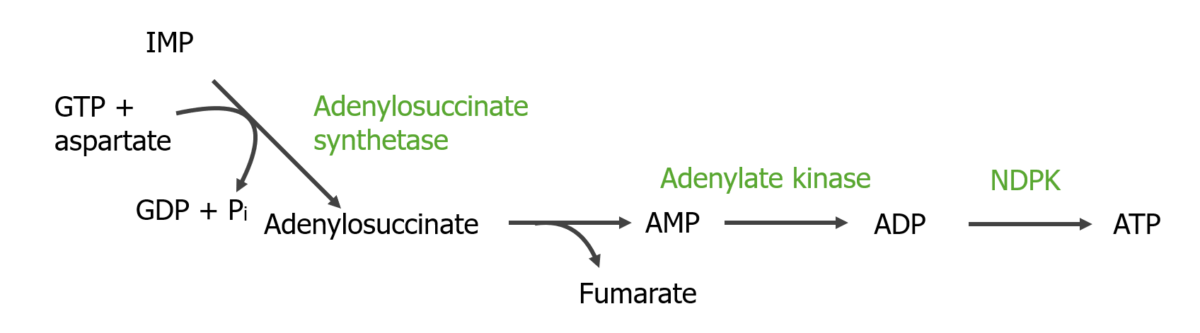
Conversión de IMP a AMP y luego a ATP:
NDPK: nucleósido difosfato quinasa (por sus siglas en inglés)
Pi: fosfato inorgánico
La síntesis de IMP, ATP y GTP está regulada para controlar la cantidad de nucleótidos de purina producidos.
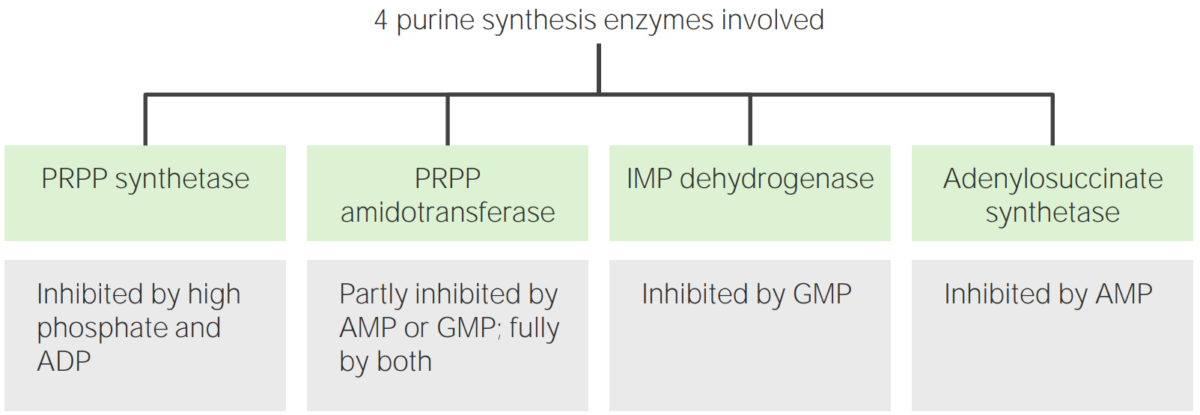
Reguladores del metabolismo de las purinas
Imagen por Lecturio.El ácido nucleico (ARN/ADN) se descompone en nucleótidos mediante nucleasas. Para degradar los nucleótidos de purina, primero se eliminan el fosfato y la ribosa, y otras reacciones conducen a la xantina y luego al ácido úrico.
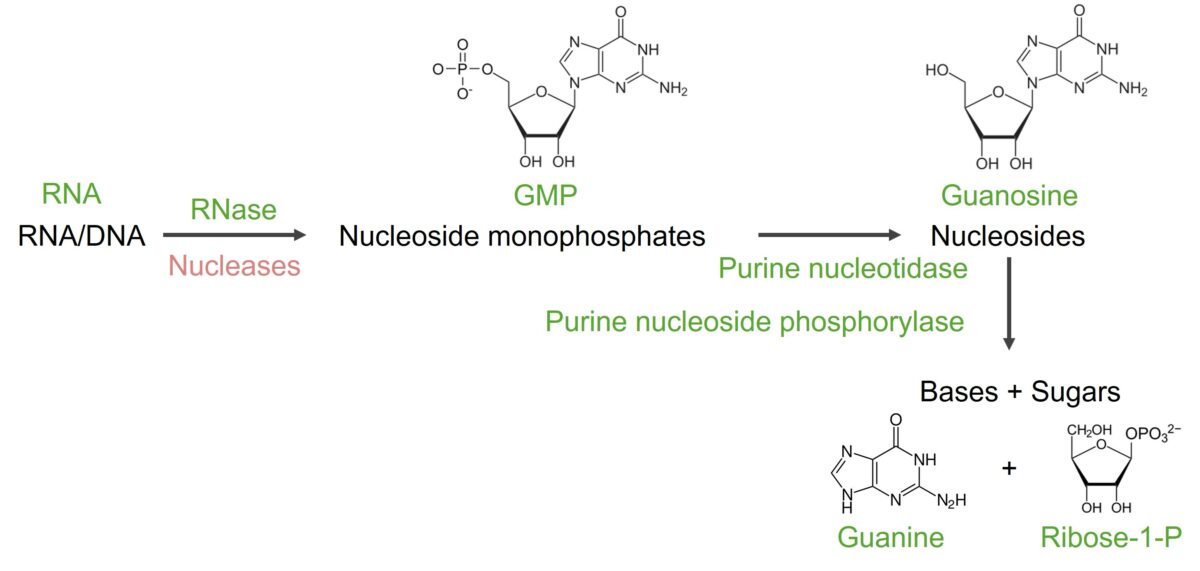
Degradación de guanina
Imagen por Lecturio.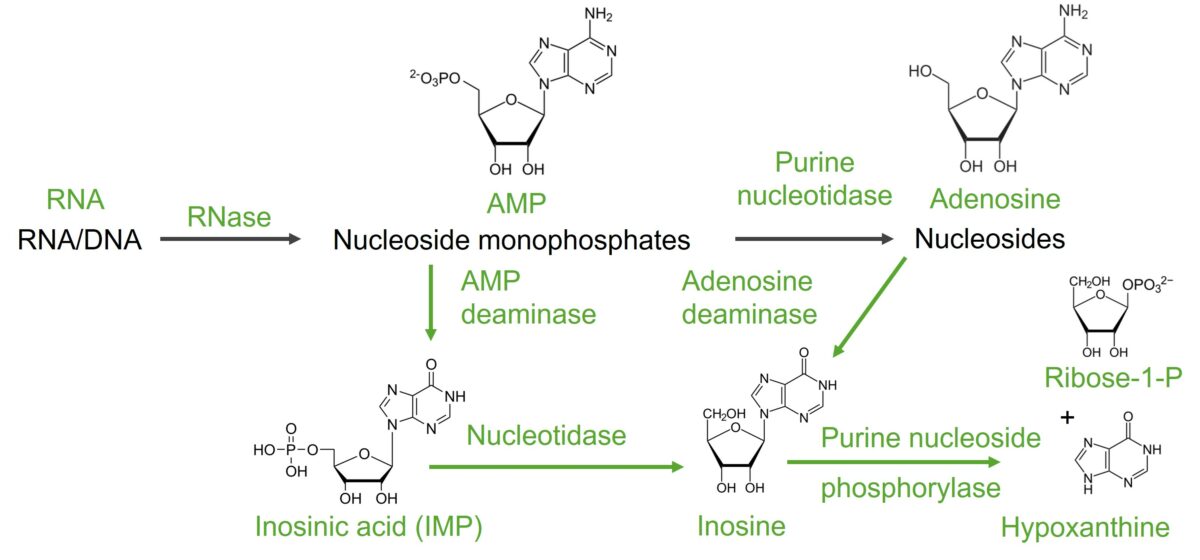
Degradación de la adenina
Imagen por Lecturio.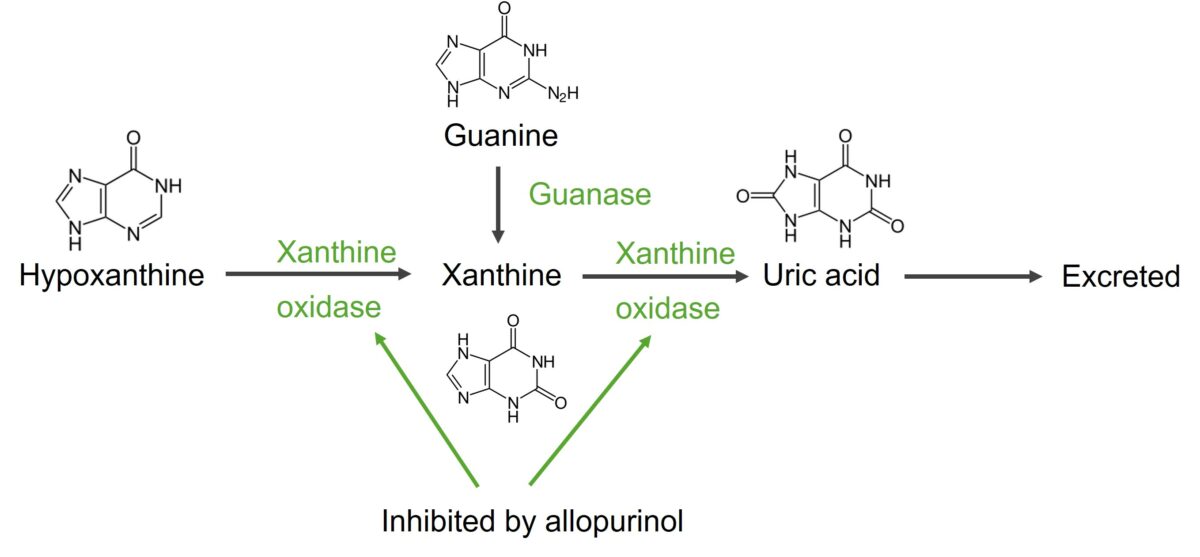
Degradación de guanina e hipoxantina en ácido úrico
Imagen por Lecturio.Síntesis de carbamoil fosfato
Síntesis de carbamoil aspartato

Reacción limitante de la síntesis de pirimidinas:
La reacción convierte al carbamoil fosfato en carbamoil aspartato, catalizada por la aspartil transcarbamoilasa (ATCase, por sus siglas en inglés). Las reacciones posteriores finalmente conducen al producto final, la citidina trifosfato (CTP). La aspartil transcarbamoilasa es activada por ATP e inhibida por CTP.
Formación del anillo de pirimidina
Oxidación del dihidroorotato
Formación de orotidina-5-monofosfato (OMP)
Descarboxilación para formar monofosfato de uridina (UMP)
Nota: las últimas 2 enzimas de esta vía, la orotato fosforribosiltransferasa y la OMP descarboxilasa, se encuentran en el mismo polipéptido, UMP sintasa. La UMP sintasa cataliza la conversión del ácido orótico en UMP.
| Paso | Enzima | Producto |
|---|---|---|
| 1 | Carbamoil fosfato sintetasa II | Carbamoil fosfato |
| 2 | Aspartil transcarbamoilasa* | Carbamoil aspartato |
| 3 | Dihidroorotasa | Ácido dihidroorótico |
| 4 | Dihidroorotato deshidrogenasa | Ácido orótico |
| 5 | Orotato fosforribosiltransferasa | OMP |
| 6 | OMP descarboxilasa | Uridina monofosfato |
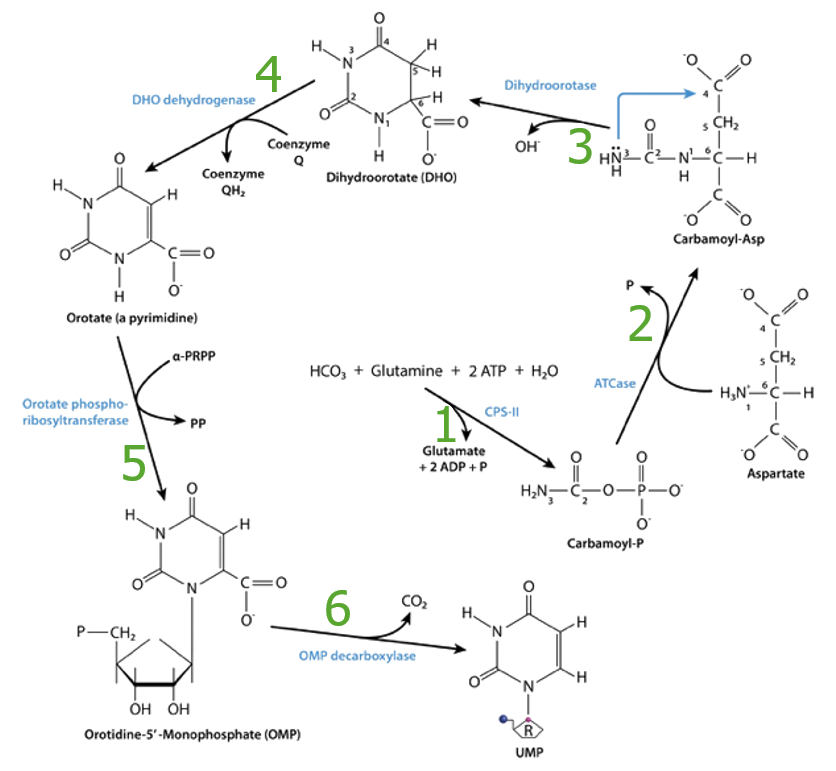
Resumen de la síntesis de pirimidinas, enzimas:
1. CPS II: carbamoil fosfato sintetasa II
2. ATCase: aspartil transcarbamoilasa (por sus siglas en inglés)
3. Dihidroorotasa
4. Dihidroorotato (DHO) deshidrogenasa
5. Orotato fosforribosiltransferasa
6. Orotidina-5-monofosfato (OMP) descarboxilasa
El UTP y el CTP se utilizan en la síntesis de ARN.
UTP:
CTP:
El ADN es diferente del ARN, ya que el ADN tiene desoxirribosa, en lugar de ribosa, y timina (5-metiluracilo), en lugar de uracilo.
Los desoxirribonucleótidos se generan a partir de sus correspondientes ribonucleótidos.
La timina es una pirimidina presente en el ADN; por lo tanto, la ribosa del nucleótido correspondiente requiere reducción.
Correlación clínica: 5-fluorouracilo: agente antimetabolito (usado en el cáncer) que inhibe la timidilato sintasa y disminuye la síntesis de ADN
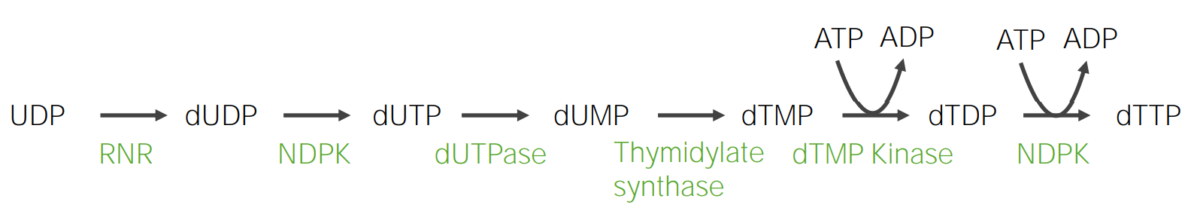
Formación de timina en forma de desoxitimidina trifosfato (dTTP)
dTDP: desoxitimidina difosfato
dTMP: desoxitimidina monofosfato
dTTP: desoxitimidina trifosfato
dUDP: desoxiuridina difosfato
dUMP: desoxiuridina monofosfato
dUTPasa: desoxiuridina trifosfatasa
NDPK: nucleósido difosfato quinasa (por sus siglas en inglés)
RNR: ribonucleótido reductasa
UDP: difosfato de uridina
Las células animales descomponen los nucleótidos de pirimidinas en bases nitrogenadas, y el uracilo y la timina resultantes se degradan (a través de reducción) en el hígado.
| Trastorno | Enzima defectuosa | Naturaleza del defecto | Manifestaciones |
|---|---|---|---|
| Hiperuricemia/gota |
|
↑ Ácido úrico | Articulaciones inflamadas y dolorosas |
| Síndrome de Lesch-Nyhan | ↓ HGPRT | Falta de enzima → defectos en la vía de reciclaje de purinas |
|
| SCID | ↓ ADA | Falta de enzima → ↓ células inmunitarias |
|
| Litiasis renal | ↓ APRT | Mutación autosómica recesiva → defectos en la vía de reciclaje de purinas |
|
| Xantinuria | ↓ Xantina oxidasa | Hipouricemia |
|
| Trastorno | Enzima defectuosa | Manifestaciones |
|---|---|---|
| Aciduria orótica |
|
|
| Aciduria orótica inducida por medicamentos | OMP descarboxilasa |
|