Los inhibidores enzimáticos se unen a las enzimas y disminuyen su actividad. Los activadores enzimáticos se unen a las enzimas y aumentan su actividad. Las moléculas que disminuyen la actividad catalítica de las enzimas pueden presentarse de diversas formas e incluyen la inhibición reversible o irreversible. La inhibición reversible puede ser competitiva, no competitiva o acompetitiva.
Última actualización: Abr 17, 2025
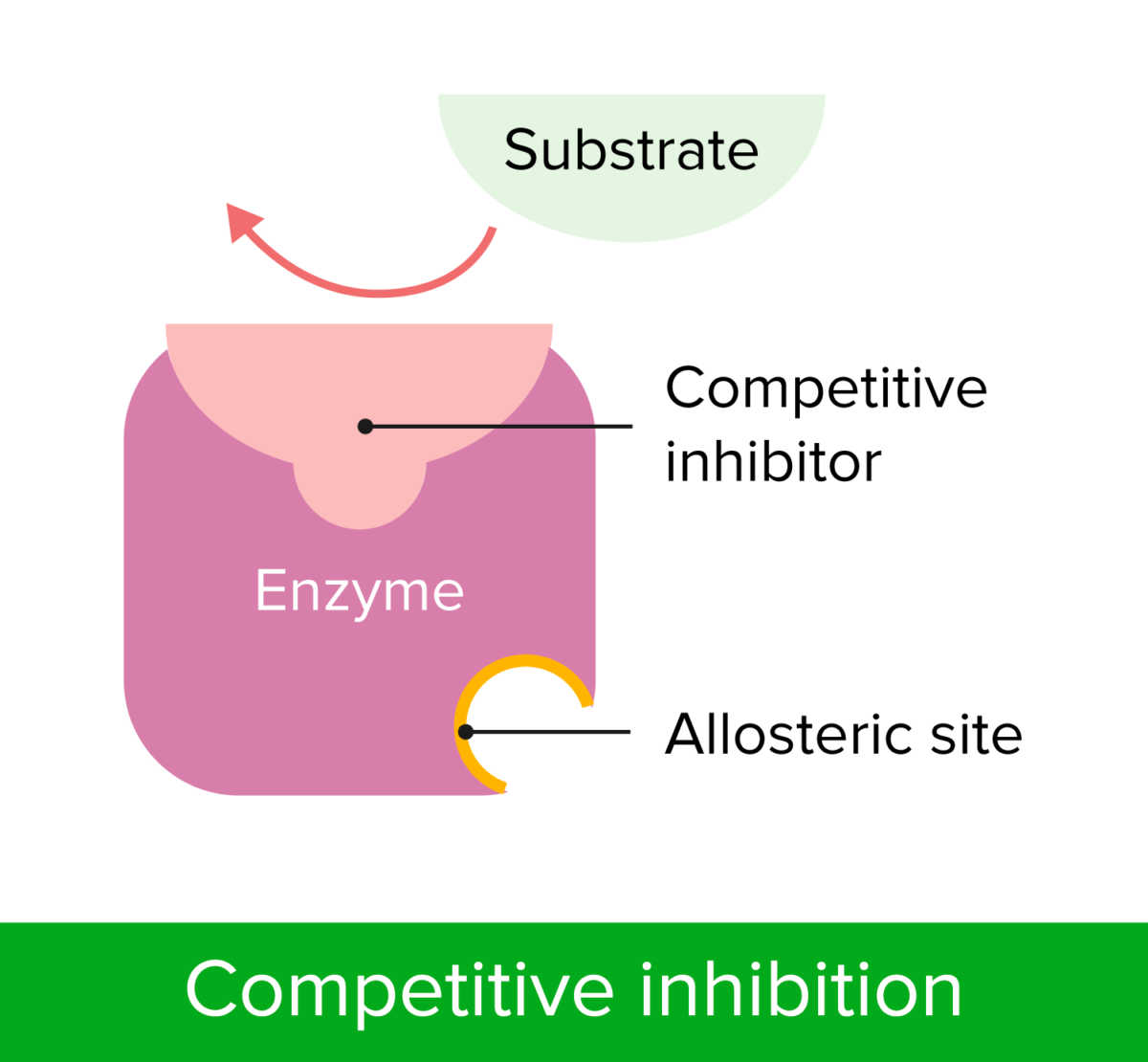
Inhibición competitiva
Imagen por Lecturio.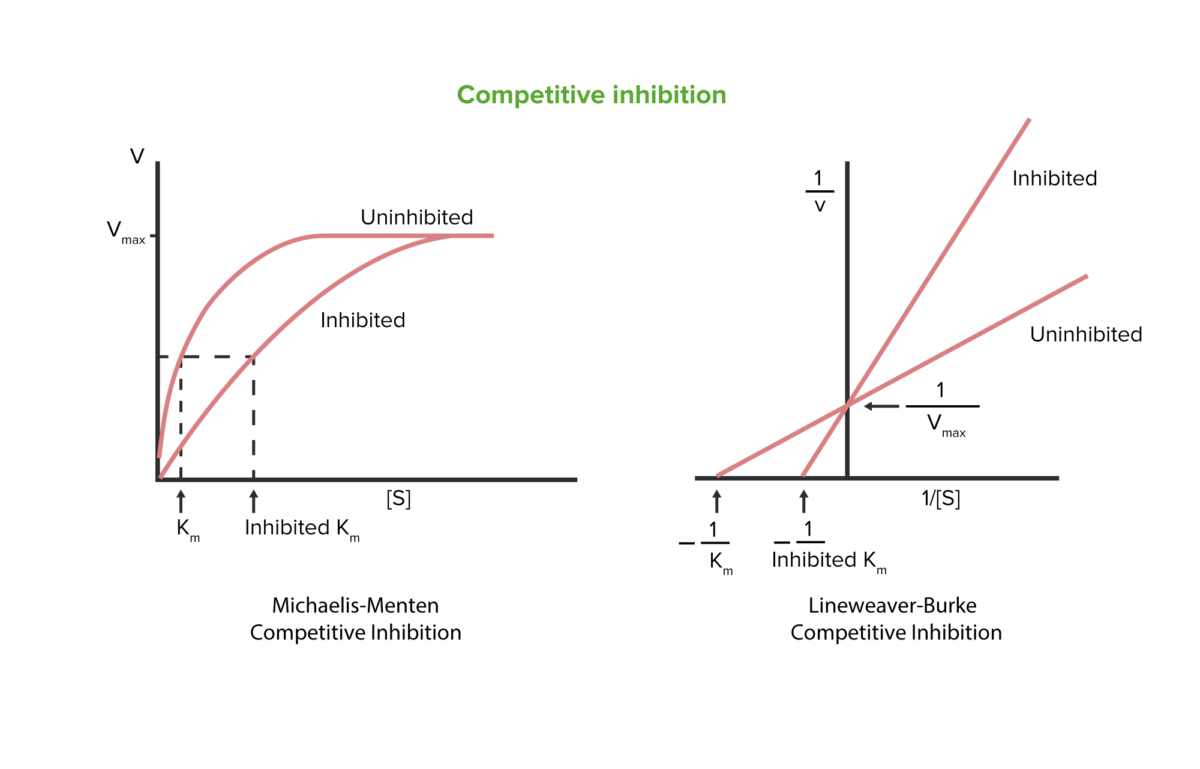
Inhibición competitiva
Imagen por Lecturio.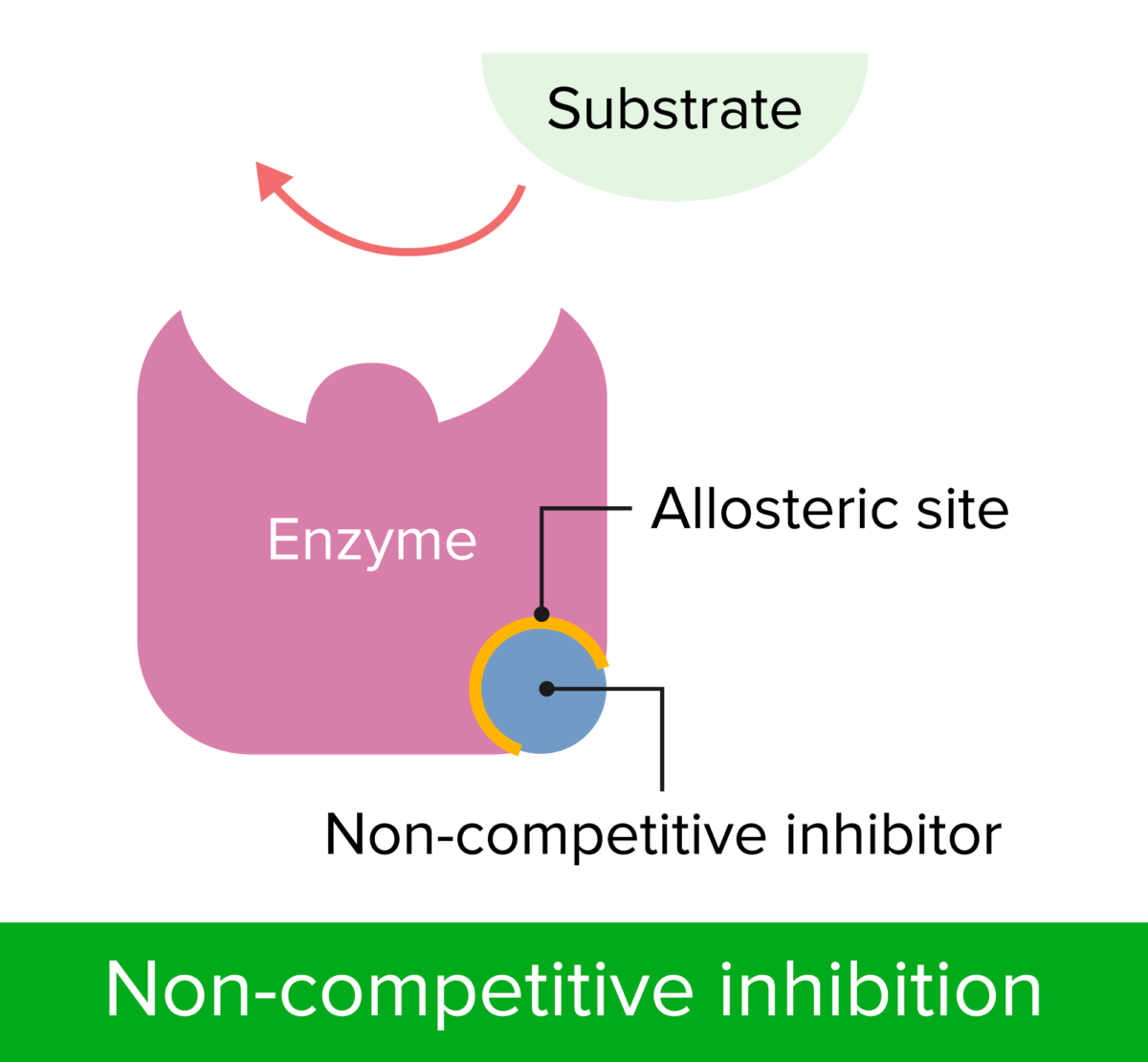
Inhibición no competitiva
Imagen por Lecturio.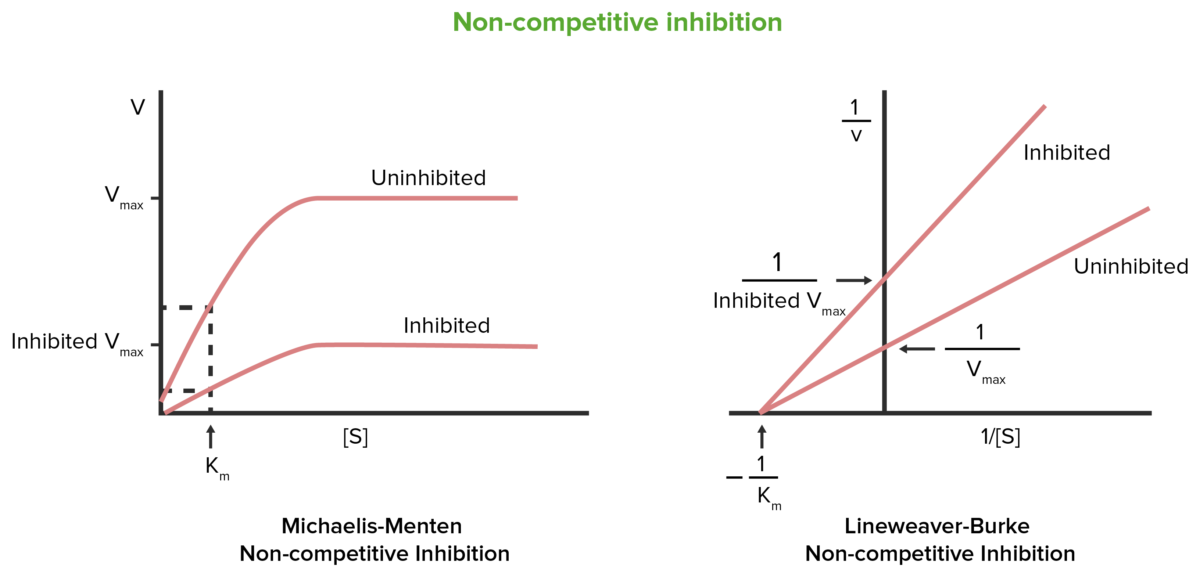
Inhibición no competitiva
Imagen por Lecturio.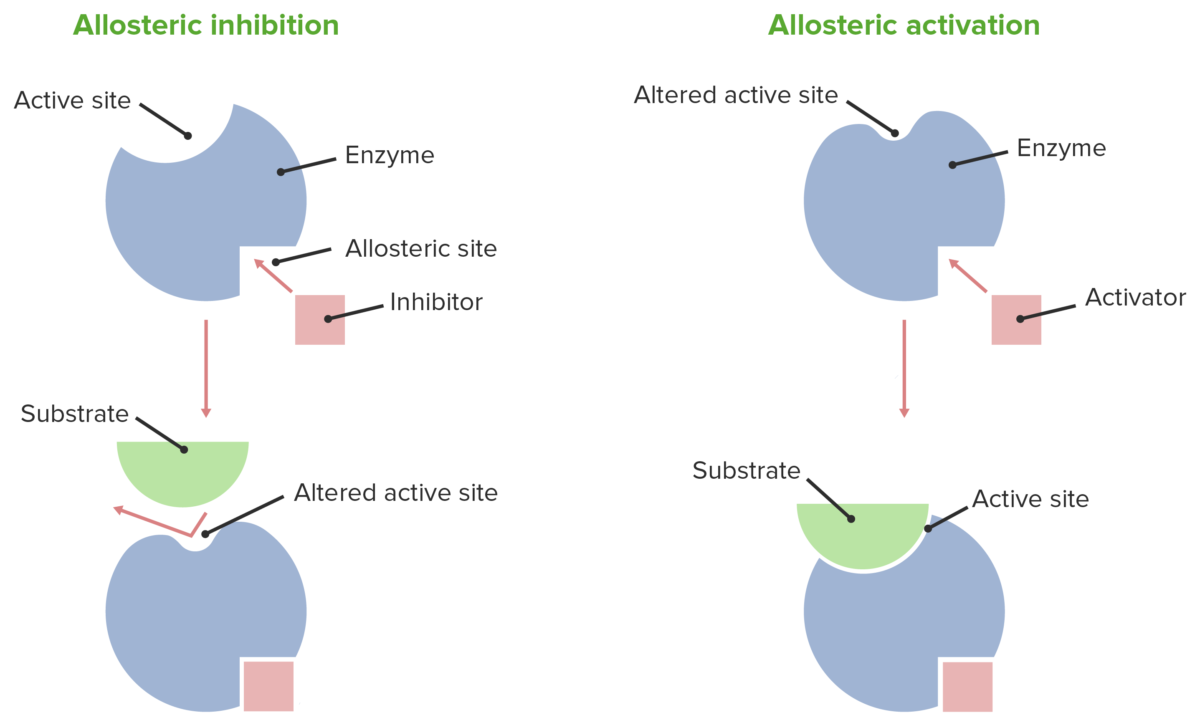
Efectos alostéricos
Imagen por Lecturio.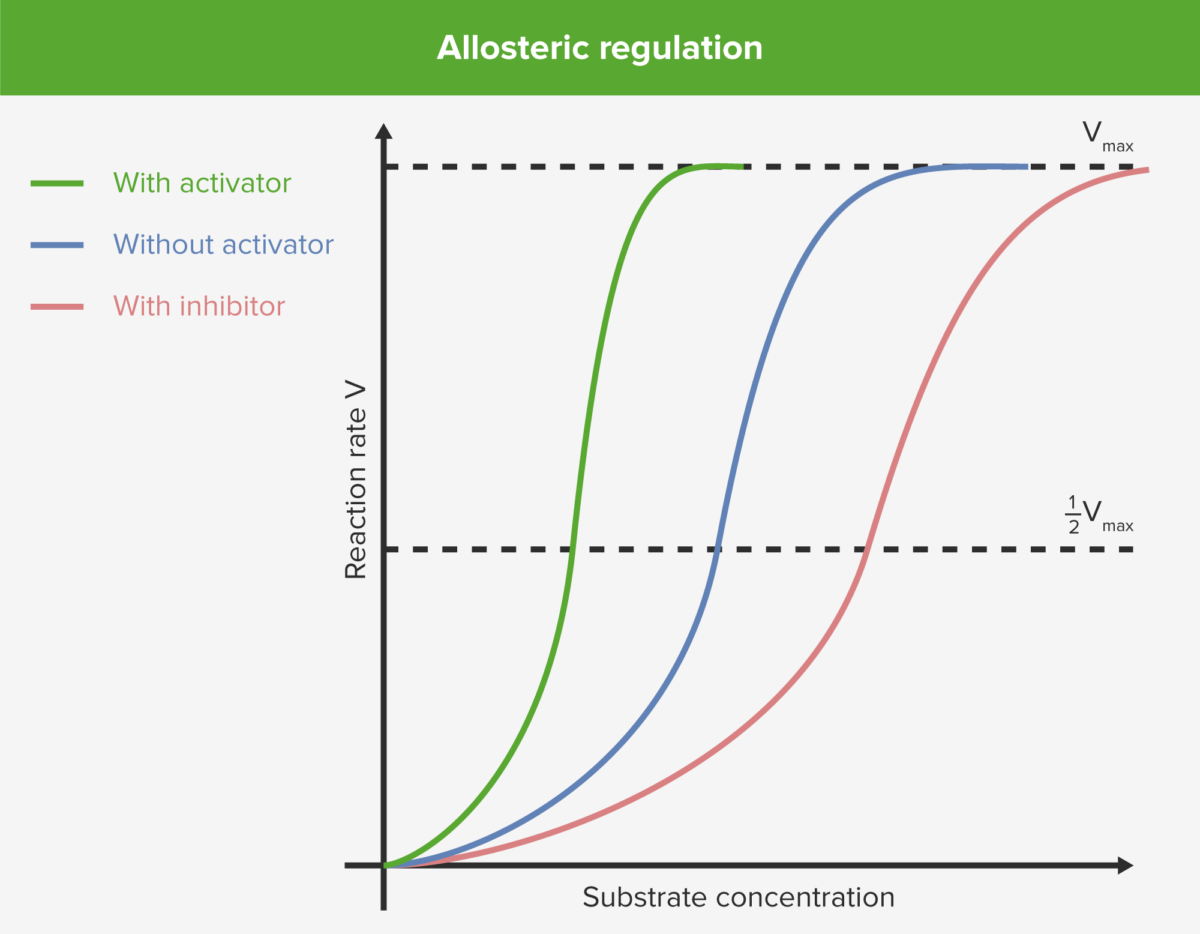
Regulación alostérica
Imagen por Lecturio.